Kaggle挑戦前時点でのデータ分析手法
はじめに
最近、重い腰を上げ、ようやくKaggleを始めました。
タイタニックやインターン限定のコンペ等には参加したことがありましたが、賞金が発生するようなKaggleに参加したことは、今までありませんでした。
データサイエンス及びエンジニアリングのスキルは研究メインで勉強している現状です。
そんな自分が、現時点でデータを与えられた場合、何から初めてどう進めるかのプロセスを本記事でまとめたいと思います。
Kaggle等進め、自分にさらに技術力がついたとき、この記事を読んで「このときはわかってなかった……」と顧みるための備忘録とも言えます。
なお、ビジネス的な話はなしとします。
データ
データはタイタニックのデータを使います。
あくまで、どういう手順で分析を進めるかに重きを置くので、精度や特徴ベクトルの有用性などは検討しません。
タイタニックの各カラムがどんなデータかは、以下を参照してください。
フォルダ構成
フォルダ構成は以下になります。
こちらを参考にしました。
各スクリプトとdataフォルダ内の関係性は次節以降で述べます。
├── data <- データ関連 │ ├── interim <- 作成途中のデータ │ ├── processed <- 学習に使うデータ │ ├── raw <- 生データ │ │ ├── test.csv │ │ ├── train.csv │ ├── submission <- 提出用データ │ │ ├── sample_submission.csv ├── scripts <- プログラム類 │ ├── models <- モデル類 │ │ ├──__init__.py │ │ ├── model.py <- モデル基底クラス │ │ ├── model_lgb.py <- LightGBMクラス │ │ ├── util.py <- 汎用クラス │ ├── generate.py <- データ作成 │ ├── analyze.py <- 分析用スクリプト │ ├── run.py <- 学習用スクリプト │ ├── config <- 汎用的処理クラス │ │ ├── features <- 特徴量のカラム群 ├── models <- 作成したモデル保存
手順
- データを作る
- データを分析する
- モデルを作り、評価する
1. データを作る
ここでの処理では、例えば、複数ファイルに別れたファイルを分析しやすいように一つのファイルにまとめるなどがあります。
タイタニックはもちろん比較的きれいなデータなので、そうはなっていません。
一見すると必要のない処理かもしれませんが、ここでは学習データとテストデータを分析するために、2つのデータを結合します。
まず、データセットを読み込み、中身を見ます。
import os import pandas as pd from IPython.core.display import display ## データセット作成 # 読込 train_df = pd.read_csv('../data/raw/train.csv') test_df = pd.read_csv('../data/raw/test.csv') display(train_df.head()) display(test_df.head())
欠損と基本的な統計値の確認をします。
# 欠損確認 display(train_df.info()) display(test_df.info()) # 統計値確認 display(train_df.describe()) display(test_df.describe())
以下は、# 欠損確認 の出力ですが、学習もテストデータもAgeとCabinが大きく欠損していることがわかります。
# 欠損確認... <class 'pandas.core.frame.DataFrame'> RangeIndex: 891 entries, 0 to 890 Data columns (total 12 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 PassengerId 891 non-null int64 1 Survived 891 non-null int64 2 Pclass 891 non-null int64 3 Name 891 non-null object 4 Sex 891 non-null object 5 Age 714 non-null float64 6 SibSp 891 non-null int64 7 Parch 891 non-null int64 8 Ticket 891 non-null object 9 Fare 891 non-null float64 10 Cabin 204 non-null object 11 Embarked 889 non-null object dtypes: float64(2), int64(5), object(5) memory usage: 83.7+ KB None <class 'pandas.core.frame.DataFrame'> RangeIndex: 418 entries, 0 to 417 Data columns (total 11 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 PassengerId 418 non-null int64 1 Pclass 418 non-null int64 2 Name 418 non-null object 3 Sex 418 non-null object 4 Age 332 non-null float64 5 SibSp 418 non-null int64 6 Parch 418 non-null int64 7 Ticket 418 non-null object 8 Fare 417 non-null float64 9 Cabin 91 non-null object 10 Embarked 418 non-null object dtypes: float64(2), int64(4), object(5) memory usage: 36.0+ KB None
学習データとテストデータを結合します。
結合するメリットはいくつかありますが、結合することで、一括にデータ補完やラベリングできることが主な理由だと思っています。
なお、テストデータには目的変数Survivedがないため、-1を補完しています。
# 結合 df = pd.concat([train_df, test_df], axis=0, ignore_index=True, sort=False) df['Survived'] = df['Survived'].fillna(-1) display(df) display(df.info()) display(df.describe())
欠損が確認された、Age、Embarked、Fareのデータを補完します。
補完の方法はいくつかありますが、今回は以下のようにしました。
- Age:年齢の平均で補完
- Embarked:最も多かったカテゴリ(港)で補完
- Fare:料金の平均で補完
# 欠損補完 df['Age'] = df['Age'].fillna(df['Age'].mean()) # 29.881137667304014 df['Embarked'] = df['Embarked'].fillna(df['Embarked'].value_counts().idxmax()) # S df['Fare'] = df['Fare'].fillna(df['Fare'].mean()) # 33.295479281345564
不必要なカラムを削除します。
本来ならば、あるカラムのデータが必要化どうかは、可視化して分析したり、モデルに入れてみて効くか見たりなどしてから、不要かどうか判断すべきです。
もし、カラムが不要であることが自明、もしくはあらかじめわかっていた場合、ここで削除する処理を入れるようにしています。
# カラム削除 df = df.drop(['Name', 'Ticket'], axis=1)
学習データとテストデータを見分けるタグ用のカラムを用意します。
Survivedの値からわかることではありますが、今回は、ブログ用にわかりやすくするため入れました。
# trainとtest df.loc[df['Survived']!=-1, 'data'] = 'train' df.loc[df['Survived']==-1, 'data'] = 'test' display(df) display(df.info()) display(df.describe())
最後に保存します。
生データから新たに生成したデータなため、中間データとしてinterimフォルダに保存しています。
# 保存 df.to_csv('../data/interim/all.csv', index=False)
2. データを分析する
ここでの処理では、機械学習の肝とも呼べる前処理をとにかく、しまくります。
実際にする大まか処理は以下になります。
- 生データの特徴量(カラム)の可視化
- 新たな特徴量作成
- 主成分分析
- クラスタリング
- 新たに作成した特徴量の可視化
今回は、可視化をメインに新たな特徴量作成は必要最低限としました。
import os import pandas as pd import numpy as np from IPython.core.display import display from sklearn.preprocessing import LabelEncoder import matplotlib.pyplot as plt import seaborn as sns from models import Util # 読込 df = pd.read_csv('../data/interim/all.csv') df.head()
生データの特徴量(カラム)の可視化をします。
まずは、死亡者と生存者に違いが見られる特徴量を探してみることにします。
Pclass、Sex、SibSp、Parch、Embarkedについて可視化します。
# 死亡者と生存者の違い df_s = df[df['data']=='train'] cols = ['Pclass', 'Sex', 'SibSp', 'Parch', 'Embarked'] for col in cols: print(col) plt.figure(figsize=(4,3)) sns.countplot(x=col, data=df_s, hue=df_s['Survived']) plt.legend( loc='upper right') plt.show()
下図の結果から、以下のことがわかります。
- Pclass:3はチケットのクラスがLowerため、多く亡くなっている
- Sex:女性より男性の方が多く亡くなっている
- SibSpとParch:自分以外に一人か二人、家族等がいた方が生存している
- Cherbourg港だけ、生存者が多い
| 特徴量 | 棒グラフ |
|---|---|
| Pclass |  |
| Sex |  |
| SibSp |  |
| Parch |  |
| Embarkd |  |
新たに特徴量を作成します。
カラムSibSpとParchから、家族の人数という新しい特徴量を作ります。
これは、SibSp – タイタニックに同乗している兄弟/配偶者の数、parch – タイタニックに同乗している親/子供の数という2つの特徴を1つにまとめる処理ともいえます。
# 家族人数 df['Family'] = df['SibSp'] + df['Parch']
また、機械学習するにはカテゴリ変数を数値に変える必要があります。
これは、以前こちらの記事でも書きました。よければ参考にしてください。
Sex、Embarked、Cabinに関して、ラベルエンコーディングをします。
なお、Cabinのみ文字の頭だけ抽出する処理をしています。
# ラベルエンコーディング lenc = LabelEncoder() lenc.fit(df['Sex']) df['Sex'] = pd.DataFrame(lenc.transform(df['Sex'])) lenc.fit(df['Embarked']) df['Embarked'] = pd.DataFrame(lenc.transform(df['Embarked'])) df['Cabin'] = df['Cabin'].apply(lambda x:str(x)[0]) lenc.fit(df['Cabin']) df['Cabin'] = pd.DataFrame(lenc.transform(df['Cabin']))
作成した新たな特徴量を可視化します。
コードはcols以外変わりません(関数にしていないのはご愛嬌ということで)。
なお、SexとEmbarkedは、先ほど可視化しているので省略しています。
# 死亡者と生存者の違い df_s = df[df['data']=='train'] cols = ['Family', 'Cabin'] for col in cols: print(col) plt.figure(figsize=(4,3)) sns.countplot(x=col, data=df_s, hue=df_s['Survived']) plt.legend( loc='upper right') plt.show()
下図の結果から、以下のことがわかります。
- Family:家族が1人~3人いる人は生存している可能性が高い
- Cabin:欠損値になっている人がかなり亡くなっている。また、1~5(B~F)は生存者の方が多い
| 特徴量 | 棒グラフ |
|---|---|
| Family |  |
| Cabin |  |
作られたデータフレームを保存します。
中間データから作成したものの内、そのまま学習データになるものはprocessedフォルダに保存しています。
中間データから中間データを作成することも当然あり、その場合は別の中間データとしてinterimフォルダに保存します。
今回は前者です。
# 保存 df.to_csv('../data/interim/all.csv', index=False)
最後に特徴量をダンプしておきます。
util.py含め、modelsフォルダ内のコードはAppendixを見てください。
# 特徴量 df = df.drop(['PassengerId', 'data', 'Survived'], axis=1) # 特徴量保存 Util.dump(df.columns, 'config/features/all.pkl')
3. モデルを作り、評価する
交差検証による学習する処理と、与えられた全データから学習する処理を2パターン書いてます。
交差検証するときは全体学習をコメントアウト、全体学習するときは交差検証をコメントアウトという使い勝手の悪さが目立ちますね。
今回は、モデルはLightGBMを用い、交差検証しているもののパラメータチューニング等はしていません。
import pandas as pd import numpy as np import matplotlib.pyplot as plt import matplotlib.pyplot as plt from models import ModelLGB, Util from sklearn.model_selection import StratifiedKFold from sklearn.metrics import classification_report, confusion_matrix, accuracy_score import lightgbm as lgb # LightGBM def run_lgb(tr_x, tr_y, te_x, te_y, run_fold_name, params, load_path=None): lgbm = ModelLGB(run_fold_name, params) # 学習 if load_path is None: build_lgb(tr_x, tr_y, lgbm) lgbm.save_model() else: lgbm.load_model() # 予測 pred = predict_lgb(te_x, lgbm, params['objective']) # 重要度可視化 plot_lgb_importance(lgbm) # 評価 print(classification_report(te_y, pred, target_names=['0', '1'])) print(confusion_matrix(te_y, pred)) acc = accuracy_score(te_y, pred) print(acc) return acc def build_lgb(tr_x, tr_y, lgbm, issave=False): lgbm.train(tr_x, tr_y) if issave: lgbm.save_model() print('saved model') def predict_lgb(te_x, lgbm, objective): pred = lgbm.predict(te_x) if objective == 'multiclass': pred = np.argmax(pred, axis=1) elif objective == 'binary': pred = [1 if p > 0.5 else 0 for p in pred] return pred # 特徴量の重要度を確認 def plot_lgb_importance(lgbm): lgb.plot_importance(lgbm.model, height = 0.5, figsize = (4,8)) plt.show() def run_cv(train_x, train_y, run_name, params): i = 0 scores = [] skf = StratifiedKFold(n_splits=3, shuffle=True, random_state=2021) for tr_idx, va_idx in skf.split(train_x, train_y): run_fold_name = f'{run_name}-{i}' tr_x, tr_y = train_x.iloc[tr_idx], train_y.iloc[tr_idx] va_x, va_y = train_x.iloc[va_idx], train_y.iloc[va_idx] score = run_lgb(tr_x, tr_y, va_x, va_y, run_fold_name, params, load_path=None) scores.append(score) i+=1 return np.array(scores) def main(): features = Util.load('config/features/all.pkl') # データ取得 df = pd.read_csv('../data/processed/all.csv') train_df = df[df['data']=='train'].reset_index(drop=True) test_df = df[df['data']=='test'].reset_index(drop=True) train_x = train_df[features] train_y = train_df['Survived'] test_x = test_df[features] # LightGBM run_name = 'lgb' params = { 'objective' : 'binary', 'metric' : 'binary_logloss', 'verbosity' : -1 } scores = run_cv(train_x, train_y, run_name, params) print(scores) print(scores.mean()) # 全体で再度学習 run_fold_name = f'{run_name}-all' lgbm_all = ModelLGB(run_fold_name, params) build_lgb(train_x, train_y, lgbm_all) pred = predict_lgb(test_x, lgbm_all, params['objective']) plot_lgb_importance(lgbm_all) left = df.loc[df['data']=='test', 'PassengerId'].reset_index(drop=True) right = pd.DataFrame(pred, columns=['Survived']) sub_df = pd.concat([left, right], axis=1) print(sub_df) sub_df.to_csv(f'../data/submission/{run_fold_name}.csv', index=False) if __name__ == "__main__": main()
交差検証の結果は以下のようになりました。
- 1回目精度:0.78451
- 2回目精度:0.84511
- 3回目精度:0.78451
- 3回の平均精度:0.80471
Kaggle側の提出結果が以下になります。

おわりに
枠組みを作りたかったですが、やる前に作るのはやはり難しいですね。
こんなのが欲しいなと思った時、随時自作して行こうと思います。
今後作りたいものとしては以下のようなものがあります。
- 学習ログの記録
- 学習ログと特徴量とモデルの関連付け(対応関係がわけわからなくなりそうなため)
- 学習終了後に通知してくれるBotの作成(学習に時間がかかる可能性があるため)
- 交差検証・パラメータチューニング用のクラス
Appendix
こちらを参考にしました。
model.pyとutil.pyは上記リンクと同じです。
ただし、util.pyに関してはUtilクラスしか、本記事では用いてません(そのため、必要に応じてコメントアウトするところがあるはず)。
init.py
from .model import Model from .model_lgb import ModelLGB from .util import Util
model_lgb.py
import os import lightgbm as lgb from .model import Model from .util import Util # LightGBM class ModelLGB(Model): def train(self, tr_x, tr_y, va_x=None, va_y=None): # データのセット isvalid = va_x is not None lgb_train = lgb.Dataset(tr_x, tr_y) if isvalid: lgb_valid = lgb.Dataset(va_x, va_y) # ハイパーパラメータの設定 params = dict(self.params) # num_round = params.pop('num_round') # 学習 if isvalid: self.model = lgb.train(params, lgb_train, valid_sets=lgb_valid) else: self.model = lgb.train(params, lgb_train) def predict(self, te_x): return self.model.predict(te_x, num_iteration=self.model.best_iteration) def save_model(self): model_path = os.path.join('../models/lgb', f'{self.run_fold_name}.model') os.makedirs(os.path.dirname(model_path), exist_ok=True) Util.dump(self.model, model_path) def load_model(self): model_path = os.path.join('../models/lgb', f'{self.run_fold_name}.model') self.model = Util.load(model_path)
参考文献
Titanic - Machine Learning from Disaster | Kaggle
タイタニック号の乗客の生存予測〜80%以上の予測精度を超える方法(探索的データ解析編) │ キヨシの命題
愛(AI)の力でパイモンを救いたい
ネタ回です。
前回の投稿から少し開きました。 就活と論文のダブルパンチで死んでたわけですが、また引き続き頑張ります。
はじめにのはじめに
本記事で出てくる画像は以下から引用してます。
株式会社miHoYo corp.mihoyo.co.jp
Bossard, Lukas and Guillaumin, Matthieu and Van Gool, Luc, Food-101 – Mining Discriminative Components with Random Forests, European Conference on Computer Vision, 2014. data.vision.ee.ethz.ch
はじめに
今回の内容は、前回の投稿内容の続き的な立ち位置になります。
趣旨
原神というゲームがあります。
最近、週一でやっているかどうかくらいのペースでしかやっていなかったります。
ぶっちゃけ、もう飽きてる節がありますが、たまに惰性でやってます。
本記事の内容は、タイトルで察しってください、と言いたいところですが趣旨を説明します。
ゲームに限りませんが、序盤から主人公と一緒にいて、何かと手助けしてくれるマスコット的なキャラクターがいるかと思います。
原神では、それがパイモンです。
ただ、主人公との出会いが釣りをしてたら釣れたという経緯があり、公式公認で非常食扱いされてます。

かわいそうなので、愛(AI)の力でパイモンを救ってみよう、というわけです。
データ
画像は二種類用意します。
一種類目は、もちろんパイモンの画像です。
Twitterから主に集めた画像を顔画像だけトリミングしたものを用意してます。
二種類目は、食べ物の画像です。
これはFood-101という101種類の食べ物の画像が1000枚ずつ、合計101000枚あるデータセットになります。
これらの画像を学習し、分類するモデルを作ることが本記事のゴールです。
環境
| 言語・ライブラリ | バージョン |
|---|---|
| python | 3.7.9 |
| pandas | 1.2.0 |
| numpy | 1.19.2 |
| scikit-learn | 0.23.2 |
| tensorflow | 2.0.0 |
なお、学習にはGPU(GTX1060 6GB)使っています。
いざ、救う
二クラス分類プログラム
pathは任意
import glob as gb import pandas as pd import numpy as np import matplotlib.pyplot as plt import seaborn as sns import tensorflow.keras as keras from tensorflow.keras.models import Sequential from tensorflow.keras.layers import Dense, Conv2D, MaxPooling2D, Flatten from tensorflow.keras.callbacks import EarlyStopping from sklearn.model_selection import train_test_split from sklearn.metrics import classification_report, confusion_matrix from sklearn.model_selection import StratifiedKFold #TensorFlowがGPUを認識しているか確認 from tensorflow.python.client import device_lib # 画像の読み込み def load_image_npy(load_path, isdir=False): if isdir: return np.array([np.load(path, allow_pickle=True) for path in gb.glob(load_path)]) else: return np.load(load_path, allow_pickle=True) # 学習データとテストデータにわける def make_train_test_data(image1, image2): X = np.concatenate([image1, image2]) y = np.array([0] * len(image1) + [1] * len(image2)) # face:0, food:1 X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.25, shuffle=True, random_state=2021) return X_train, X_test, y_train, y_test # モデル構築 def build_cnn_model(): model = Sequential() # 入力画像 64x64x3 (縦の画素数)x(横の画素数)x(チャンネル数) model.add(Conv2D(16, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal', input_shape=(64, 64, 3))) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Conv2D(64, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal')) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Flatten()) model.add(Dense(1, activation='sigmoid')) model.compile( loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'] ) model.summary() return model def learn_model(model, X_train, y_train): X_train, X_val, y_train, y_val = train_test_split(X_train, y_train, test_size=0.25, shuffle=True, random_state=2021) early_stopping = EarlyStopping(monitor='val_loss', min_delta=1.0e-3, patience=20, verbose=1) hist = model.fit(X_train, y_train, batch_size=1000, verbose=2, epochs=100, validation_data=(X_val, y_val), callbacks=[early_stopping]) return hist def evaluate_model(model, X_test, y_test): score = model.evaluate(X_test, y_test, verbose=0) print('Test loss:', score[0]) print('Test accuracy:', score[1]) return score def predict_model(model, X_test): pred_y = model.predict(X_test, batch_size=128) # pred_y = np.argmax(pred_y, axis=1) return pred_y def save_model(path, model): model.save(path) print('saved model: ', path) # 評価系のグラフをプロット def plot_evaluation(eval_dict, key1, key2, ylabel, save_path=None): plt.figure(figsize=(10,7)) plt.plot(eval_dict[key1], label=key1) plt.plot(eval_dict[key2], label=key2) plt.ylabel(ylabel) plt.xlabel('epoch') plt.legend() if save_path is None: plt.show() else: plt.savefig(save_path) def plot_cmx_heatmap(cmx, labels, save_path=None): df_cmx = pd.DataFrame(cmx, index=labels, columns=labels) plt.figure(figsize=(10,7)) sns.heatmap(df_cmx, annot=True, fmt='d') plt.ylim(0, len(labels)+1) if save_path is None: plt.show() else: plt.savefig(save_path) def main(): # GPUの動作確認 # print(device_lib.list_local_devices()) labels = ['food', 'face'] save_file_name = 'bin.png' face_image = load_image_npy('D:/Illust/Paimon/interim/npy_face_only/paimon_face_augmentation.npy') food_image = load_image_npy('D:/OpenData/food-101/interim/npy_food-101.npy') food_image = food_image[np.random.choice(food_image.shape[0], 10000, replace=False), :] # food_image101000枚の画像からランダムに10000枚抽出 print(face_image.shape) print(food_image.shape) X_train, X_test, y_train, y_test = make_train_test_data(face_image, food_image) score_list = [] kf = StratifiedKFold(n_splits=3, shuffle=True, random_state=2021) for train_idx, val_idx in kf.split(X_train, y_train): train_x, val_x = X_train[train_idx], X_train[val_idx] train_y, val_y = y_train[train_idx], y_train[val_idx] model = build_cnn_model() hist = learn_model(model, train_x, train_y) score = evaluate_model(model, val_x, val_y) pred_y = predict_model(model, val_x) pred_y = [1 if y > 0.9 else 0 for y in pred_y.flatten()] score_list.append(score[1]) print(classification_report(val_y, pred_y, target_names=labels)) cmx = confusion_matrix(val_y, pred_y) print(cmx) # plot_cmx_heatmap(cmx, labels) print(score_list) print(np.array(score_list).mean()) # 再度学習 model = build_cnn_model() hist = learn_model(model, X_train, y_train) plot_evaluation(hist.history, 'loss', 'val_loss', 'loss', 'figures/loss_'+save_file_name) plot_evaluation(hist.history, 'accuracy', 'val_accuracy', 'accuracy', 'figures/acc_'+save_file_name) y_pred = predict_model(model, X_test) y_pred = [1 if y > 0.9 else 0 for y in y_pred.flatten()] print(classification_report(y_test, y_pred, target_names=labels)) cmx = confusion_matrix(y_test, y_pred) print(cmx) plot_cmx_heatmap(cmx, labels, save_path='figures/cmx_'+save_file_name) if __name__ == "__main__": main()
全体の流れ
1. 画像読み込み
- パイモン画像:8338枚(オーグメンテーション済み)
- 食べ物の画像:10000枚
2. 学習データとテストデータに分割
学習するための学習データと最終的に評価するテストデータにわけます(全体データ数の25%をテストデータに)
このとき、正解ラベルも加えます。
3. 交差検証
学習データの中から3分割して交差検証します。
予測値は0~1の範囲で出力されます。0に近いほどパイモン、1に近いほど食べ物の画像となります。予測値から0.9より大きな値は1、0.9以下の値は0となるようにしました。
精度と損失の学習曲線を可視化し、適合率、再現率、F値、精度、混同行列により定量的にモデルを評価します。
このとき、3分割した平均精度も算出します
4. 学習データ全体で学習
本来なら交差検証でチューニング等済ませてから全体で学習しますが、今回は交差検証後にそのまま全体で学習してます。
学習が高速に行えたので、どのくらい精度にブレがあるか調べたかったため交差検証しているだけです。
5. 結果を出力・保存
こちらも交差検証同様、学習データ全体の精度と損失の学習曲線を可視化し、 学習データ全体の適合率、再現率、F値、精度、混同行列により定量的にモデルを評価します。
パイモン画像のオーグメンテーション
4000枚ほどの画像を約2倍の8000枚ほどまで水増ししました。 詳しいオーグメンテーションのコードは後述します。
以下、行ったオーグメンテーション
- ランダムに±指定した角度の範囲で回転
- ランダムに±指定した横幅に対する割合の範囲で左右方向移動
- ランダムに±指定した縦幅に対する割合の範囲で左右方向移動
- ランダムにズームする
- 水平方向に入力をランダムに反転
- 垂直方向に入力をランダムに反転
これらの処理をあらかじめ行っておき、保存したものを最初に読み込んでます。
二クラス分類結果
かなり、見にくいですが……。
学習曲線は学習中の検証データに対する精度と損失です。
最終的なテストデータに対する評価が混同行列になります。
| 学習曲線(精度) | 学習曲線(損失) | 混同行列 |
|---|---|---|
 |
 |
 |
パイモンは食べ物(非常食)でないことを100%分類できました!
これは完全に救ってしまったのではなかろうか……
と、言いたいところですが交差検証評価するデータを変えると100%でないときがあります(Appendix参照)。
加えてCNNも対して工夫していないにも関わらず、この高い精度です。
つまり、ぶっちゃけ機械学習のタスクとして二クラス(パイモンと食べ物)を分類することは、そう難しくありません。
実はここまでコードを書くのは割と一瞬だったので、もう少し難しいタスクに挑戦します。
多クラス分類プログラム
ここからが本番です。
二クラス分類プログラムと同じ関数等ありますが、すべて掲載します。
import os import glob as gb import pandas as pd import numpy as np import cv2 import seaborn as sns import matplotlib.pyplot as plt import re import tensorflow.keras as keras from tensorflow.keras.models import Sequential, Model from tensorflow.keras.layers import AveragePooling2D, Dense, Conv2D, MaxPooling2D, Flatten, Input, Activation, add, Add, Dropout, BatchNormalization from tensorflow.keras.optimizers import SGD from tensorflow.keras.callbacks import EarlyStopping from tensorflow.keras.utils import to_categorical from sklearn.model_selection import train_test_split from sklearn.metrics import classification_report, confusion_matrix from sklearn.metrics import accuracy_score from sklearn.metrics import recall_score from sklearn.metrics import precision_score from sklearn.metrics import f1_score from sklearn.model_selection import StratifiedKFold from tensorflow.keras.models import load_model from tensorflow.keras.applications import InceptionV3 from tensorflow.keras.preprocessing.image import ImageDataGenerator, load_img, save_img, img_to_array, array_to_img #TensorFlowがGPUを認識しているか確認 from tensorflow.python.client import device_lib def load_image_npy(load_path, isdir=False): if isdir: return np.array([np.load(path, allow_pickle=True) for path in gb.glob(load_path)]) else: return np.load(load_path, allow_pickle=True) def make_train_test_data(image1, image2, labels): X = np.concatenate([image1, image2], axis=0) label_list = [0] * len(image1) for i in range(len(labels)): label_list += [i+1] * 1000 y = np.array(label_list) X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.25, shuffle=True, random_state=2021) return X_train, X_test, y_train, y_test # モデル構築 def build_cnn_model(): model = Sequential() # 入力画像 64x64x3 (縦の画素数)x(横の画素数)x(チャンネル数) model.add(Conv2D(16, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal', input_shape=(64, 64, 3))) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Conv2D(64, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal')) model.add(MaxPooling2D(pool_size=(2, 2))) # model.add(Conv2D(256, kernel_size=(3, 3), activation='relu', kernel_initializer='he_normal')) # model.add(MaxPooling2D(pool_size=(2, 2))) # model.add(Dropout(0.25)) model.add(Flatten()) # model.add(Dense(3200, activation='relu',kernel_initializer='he_normal')) # model.add(Dense(800, activation='relu', kernel_initializer='he_normal')) # model.add(Dense(120, activation='relu', kernel_initializer='he_normal')) # model.add(Dropout(0.5)) model.add(Dense(15, activation='softmax')) model.compile( loss='categorical_crossentropy', optimizer='adam', metrics=['accuracy'] ) model.summary() return model def build_imagenet(): base_model = InceptionV3(weights='imagenet', include_top=False, input_tensor=Input(shape=(128, 128, 3))) x = base_model.output x = AveragePooling2D(pool_size=(2, 2))(x) x = Dropout(.4)(x) x = Flatten()(x) predictions = Dense(15, activation='softmax')(x) model = Model(base_model.input, predictions) opt = SGD(lr=.01, momentum=.9) model.compile(optimizer=opt, loss='categorical_crossentropy', metrics=['accuracy']) model.summary() return model def learn_model(model, X_train, y_train, X_val, y_val): early_stopping = EarlyStopping(monitor='val_loss', min_delta=1.0e-3, patience=20, verbose=1) hist = model.fit(X_train, y_train, batch_size=32, verbose=2, steps_per_epoch=X_train.shape[0] // 32, epochs=100, validation_data=(X_val, y_val), callbacks=[early_stopping] ) return hist def learn_model_generator(model, X_train, y_train, X_val, y_val, tr_datagen, va_datagen): early_stopping = EarlyStopping(monitor='val_loss', min_delta=1.0e-3, patience=20, verbose=1) hist = model.fit_generator(tr_datagen.flow(X_train, y_train, batch_size=32), verbose=2, steps_per_epoch=X_train.shape[0] // 32, epochs=100, validation_data=(X_val, y_val), callbacks=[early_stopping] ) return hist # 画像の水増し def make_datagen(rr=30, wsr=0.1, hsr=0.1, zr=0.2, val_spilit=0.2, hf=True, vf=True): datagen = ImageDataGenerator( featurewise_center = False, # データセット全体で,入力の平均を0にするかどうか samplewise_center = False, # 各サンプルの平均を0にするかどうか featurewise_std_normalization = False, # 入力をデータセットの標準偏差で正規化するかどうか samplewise_std_normalization = False, # 各入力をその標準偏差で正規化するかどうか zca_whitening = False, # ZCA白色化を適用するかどうか rotation_range = rr, # ランダムに±指定した角度の範囲で回転 width_shift_range = wsr, # ランダムに±指定した横幅に対する割合の範囲で左右方向移動 height_shift_range = hsr, # ランダムに±指定した縦幅に対する割合の範囲で左右方向移動 zoom_range = zr, # 浮動小数点数または[lower,upper].ランダムにズームする範囲.浮動小数点数が与えられた場合,[lower, upper] = [1-zoom_range, 1+zoom_range]です. horizontal_flip = hf, # 水平方向に入力をランダムに反転するかどうか vertical_flip = vf, # 垂直方向に入力をランダムに反転するかどうか validation_split = val_spilit # 検証のために予約しておく画像の割合(厳密には0から1の間) ) return datagen def evaluate_model(model, X_test, y_test): score = model.evaluate(X_test, y_test, verbose=0) print('Test loss:', score[0]) print('Test accuracy:', score[1]) return score def predict_model(model, X_test): y_pred = model.predict(X_test, batch_size=128) # y_pred = np.argmax(y_pred, axis=1) return y_pred def save_model(path, model): model.save(path) print('saved model: ', path) # 評価系のグラフをプロット def plot_evaluation(eval_dict, key1, key2, ylabel, save_path=None): plt.figure(figsize=(10,7)) plt.plot(eval_dict[key1], label=key1) plt.plot(eval_dict[key2], label=key2) plt.ylabel(ylabel) plt.xlabel('epoch') plt.legend() if save_path is None: plt.show() else: plt.savefig(save_path) def main(): # GPUの動作確認 # print(device_lib.list_local_devices()) face_images = load_image_npy('D:/Illust/Paimon/interim/npy_face_only/paimon_face.npy') food_images = load_image_npy('D:/OpenData/food-101/interim/npy_food-101_64/npy_food-101_64.npy') # print(face_images) # print(food_images) print(face_images.shape) print(food_images.shape) # Food-101のデータがディレクトリでわけられているのでディレクトリ名=ラベルとしている labels = [re.split('[\\\.]',path)[-2] for path in gb.glob('D:/OpenData/food-101/interim/npy_food-101_64/food/*')] X_train, X_test, y_train, y_test = make_train_test_data(face_images, food_images, labels) X_train, X_val, y_train, y_val = train_test_split(X_train, y_train, test_size=0.2, shuffle=True, random_state=2021) labels = ['paimon'] + labels # ラベルをOne-Hotに変換 y_train = to_categorical(y_train) y_val = to_categorical(y_val) print(X_train.shape) print(X_val.shape) print(X_test.shape) print(y_train.shape) print(y_val.shape) print(y_test.shape) # 学習 model = build_cnn_model() # model = build_imagenet() hist = learn_model(model, X_train, y_train, X_val, y_val) # train_datagen = make_datagen() # valid_datagen = ImageDataGenerator() # hist = learn_model_generator(model, X_train, y_train, X_val, y_val, train_datagen, valid_datagen) save_file_name = 'same_bin.png' # 実験ごとに適宜変える plot_evaluation(hist.history, 'loss', 'val_loss', 'loss', 'figures/loss_'+save_file_name) plot_evaluation(hist.history, 'accuracy', 'val_accuracy', 'accuracy', 'figures/acc_'+save_file_name) y_pred = predict_model(model, X_test) y_pred = np.argmax(y_pred, axis=1) print(classification_report(y_test, y_pred, target_names=labels)) cmx = confusion_matrix(y_test, y_pred) print(cmx) df_cmx = pd.DataFrame(cmx, index=labels, columns=labels) plt.figure(figsize=(10,7)) sns.heatmap(df_cmx, annot=True, fmt='d') plt.ylim(0, len(labels)+1) # plt.show() plt.savefig('figures/cmx_'+save_file_name) if __name__ == "__main__": main()
補足説明
二クラス分類プログラムとの変更点
多クラス分類プログラムでは、オーグメンテーションをこのプログラム内でやってます。
交差検証は一回の学習の処理が重い(長い)のでやっていないです。
多クラス分類 = 15クラス分類のモデルを作ります。
- パイモン1クラス
- 食べ物14クラス
参考までに、下表にパイモンと食べ物14種類を並べました(ツナタルタルだけよくわかりませんが)。
| food(en) | food(ja) | image |
|---|---|---|
| paimon | パイモン |  |
| baby_back_ribs | スペアリブ |  |
| cup_cakes | カップケーキ |  |
| dumplings | 小籠包 |  |
| edamame | 枝豆 |  |
| guacamole | ワカモレ |  |
| miso_soup | 味噌汁 |  |
| mussels | ムール貝 |  |
| nachos | ナチョス |  |
| oysters | 牡蠣 |  |
| pancakes | パンケーキ |  |
| ramen | ラーメン |  |
| sushi | 寿司 |  |
| tuna_tartare | ツナタルタル? |  |
| waffles | ワッフル |  |
実験概要
全部で6種類実験したので、その結果を載せます。
- 二クラス分類と同じネットワークアーキテクチャ
- 二クラス分類と同じネットワークアーキテクチャ(オーグメンテーション有り)
- CNNの層を増加
- CNNの層を増加(オーグメンテーション有り)
- Inception-v3
- Inception-v3(オーグメンテーション有り)
1. 二クラス分類と同じネットワークアーキテクチャ
上記プログラムがこれになります。
ただし、出力15クラスなので出力層のノード数を15にし、活性化関数をシグモイド関数からソフトマックス関数にしてます。
同様に多クラス分類なので、損失関数をbinary_crossentropyからcategorical_crossentropyにしてます。
それ以外は同じです。
# モデル構築 def build_cnn_model(): model = Sequential() # 入力画像 64x64x3 (縦の画素数)x(横の画素数)x(チャンネル数) model.add(Conv2D(16, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal', input_shape=(64, 64, 3))) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Conv2D(64, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal')) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Flatten()) model.add(Dense(15, activation='softmax')) # 15クラスで出力してほしいので15に、活性化関数をソフトマックス関数に model.compile( loss='categorical_crossentropy', # 多クラス分類なので損失関数はcategorical_crossentropyに optimizer='adam', metrics=['accuracy'] ) model.summary() return model
2. 二クラス分類と同じネットワークアーキテクチャ(オーグメンテーション有り)
新しく、以下の関数を定義します。
これらのコードで画像の水増し及び、学習を行います。
def learn_model_generator(model, X_train, y_train, X_val, y_val, tr_datagen, va_datagen): early_stopping = EarlyStopping(monitor='val_loss', min_delta=1.0e-3, patience=20, verbose=1) hist = model.fit_generator(tr_datagen.flow(X_train, y_train, batch_size=32), verbose=2, steps_per_epoch=X_train.shape[0] // 32, epochs=100, validation_data=(X_val, y_val), callbacks=[early_stopping] ) return hist # 画像の水増し def make_datagen(rr=30, wsr=0.1, hsr=0.1, zr=0.2, val_spilit=0.2, hf=True, vf=True): datagen = ImageDataGenerator( featurewise_center = False, # データセット全体で,入力の平均を0にするかどうか samplewise_center = False, # 各サンプルの平均を0にするかどうか featurewise_std_normalization = False, # 入力をデータセットの標準偏差で正規化するかどうか samplewise_std_normalization = False, # 各入力をその標準偏差で正規化するかどうか zca_whitening = False, # ZCA白色化を適用するかどうか rotation_range = rr, # ランダムに±指定した角度の範囲で回転 width_shift_range = wsr, # ランダムに±指定した横幅に対する割合の範囲で左右方向移動 height_shift_range = hsr, # ランダムに±指定した縦幅に対する割合の範囲で左右方向移動 zoom_range = zr, # 浮動小数点数または[lower,upper].ランダムにズームする範囲.浮動小数点数が与えられた場合,[lower, upper] = [1-zoom_range, 1+zoom_range]です. horizontal_flip = hf, # 水平方向に入力をランダムに反転するかどうか vertical_flip = vf, # 垂直方向に入力をランダムに反転するかどうか validation_split = val_spilit # 検証のために予約しておく画像の割合(厳密には0から1の間) ) return datagen
上記のコードで学習するために、main関数を書き換えます。
# before # hist = learn_model(model, X_train, y_train, X_val, y_val) # after train_datagen = make_datagen() valid_datagen = ImageDataGenerator() hist = learn_model_generator(model, X_train, y_train, X_val, y_val, train_datagen, valid_datagen)
以下、オーグメンテーション無しのときはbeforeで学習し、有りのときはafterで学習します。
3. CNNの層を増加
以下のようにbuild_cnn_model関数を書き換えます。
- もう一層分、隠れ層を追加
- 全結合層のあと、徐々に15クラスまで次元を落としていく
- 過学習を防ぐDropout層を追加
def build_cnn_model(): model = Sequential() # 入力画像 64x64x3 (縦の画素数)x(横の画素数)x(チャンネル数) model.add(Conv2D(16, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal', input_shape=(64, 64, 3))) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Conv2D(64, kernel_size=(5, 5), activation='relu', kernel_initializer='he_normal')) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Conv2D(256, kernel_size=(3, 3), activation='relu', kernel_initializer='he_normal')) model.add(MaxPooling2D(pool_size=(2, 2))) model.add(Dropout(0.25)) model.add(Flatten()) model.add(Dense(3200, activation='relu',kernel_initializer='he_normal')) model.add(Dense(800, activation='relu', kernel_initializer='he_normal')) model.add(Dense(120, activation='relu', kernel_initializer='he_normal')) model.add(Dropout(0.5)) model.add(Dense(15, activation='softmax')) model.compile( loss='categorical_crossentropy', optimizer='adam', metrics=['accuracy'] ) model.summary() return model
4. CNNの層を増加(オーグメンテーション有り)
3. CNNの層を増加のbuild_cnn_model関数でモデルを構築し、 2. 二クラス分類と同じネットワークアーキテクチャ(オーグメンテーション有り)のコードで学習します。
5. Inception-v3
Inception-v3はGoogleによって開発された、画像の1000クラス分類を行うよう学習された深層学習モデルです。
この学習済みモデルを転移学習して予測します。
↓ Inception-v3に関する論文
C. Szegedy, V. Vanhoucke, S. Ioffe, and J. Shlens. Rethinking the inception architecture for computer vision. In Proc. of CVPR, 2016.
inception-v3を用いて、food-101のデータセット101クラス分類タスクとして、精度約82%出したリポジトリが以下になります。
コード書く上で参考にさせていただきました。
build_cnn_model関数の代わりに以下の関数を定義します。
def build_imagenet(): base_model = InceptionV3(weights='imagenet', include_top=False, input_tensor=Input(shape=(128, 128, 3))) x = base_model.output x = AveragePooling2D(pool_size=(2, 2))(x) x = Dropout(.4)(x) x = Flatten()(x) predictions = Dense(15, activation='softmax')(x) model = Model(base_model.input, predictions) opt = SGD(lr=.01, momentum=.9) model.compile(optimizer=opt, loss='categorical_crossentropy', metrics=['accuracy']) model.summary() return model
今まで画像のピクセルを64x64で学習させていましたが、inception-v3では128x128で学習させます。
これは、incepiton-v3がピクセルの幅と高さを75以上にしないといけないためです。
main関数で画像を読み込んだ直後に以下のコードを入れれば、画像を128x128にリサイズできます。
できますが、かなり強引な手法なのでメモリは食いまくります。当方、16GBでギリといった感じです。
resize_num = 128 face_images = np.array([cv2.resize(face_image, (resize_num,resize_num)) for face_image in face_images]) food_images = np.array([cv2.resize(food_image, (resize_num,resize_num)) for food_image in food_images])
build_imagenet関数呼び出し時を書き換えます。
# model = build_cnn_model()
model = build_imagenet()
6. Inception-v3(オーグメンテーション有り)
5. Inception-v3のbuild_cnn_model関数でモデルを構築し、 2. 二クラス分類と同じネットワークアーキテクチャ(オーグメンテーション有り)のコードで学習します。
多クラス分類結果
- 二クラス分類と同じネットワークアーキテクチャ
- 二クラス分類と同じネットワークアーキテクチャ(オーグメンテーション有り)
- CNNの層を増加
- CNNの層を増加(オーグメンテーション有り)
- Inception-v3
- Inception-v3(オーグメンテーション有り)
| 学習曲線(精度) | 学習曲線(損失) | 混同行列 | |
|---|---|---|---|
| 1 |  |
 |
 |
| 2 |  |
 |
 |
| 3 |  |
 |
 |
| 4 |  |
 |
 |
| 5 |  |
 |
 |
| 6 |  |
 |
 |
1と2
1は精度47%でした。全体の半分以上間違ってるということになります。
2のオーグメンテーション有りにすることでなんとか、精度57%になりました。
3と4
3は1と同じ精度47%でした。
4の方は62%でした。2より少しだけ精度が上がった程度です。
今回の工夫はあまり意味なったかもしれません。
5と6
5は劇的に伸びて、精度82%まで行きました。
6はさらに伸びる! かと思いきや同じ82%でした。
しかし、パイモンに関しては適合率、再現率ともに1.0なので、誤判定も見逃しもせず判定できたことになります。1~5ではこうはなりませんでした。
つまり、パイモンだけは完全に分類しきったので、救ったと言っても過言ではないでしょう!
なお、食べ物はムール貝とスペアリブを間違えたり、パンケーキとワッフルを間違えたりしていることが多かったです。
ぷち考察
6でパイモンは完全に分類しきりましたが、食べ物同士は結構間違ってます。
Food-101のデータセットは食べ物が複数写っている画像もあります。その当たりを考慮してモデルを作成していないのが原因の一つかと思われます。
極端に言えば、人が写ってる画像もありました。
また、Food-101のデータセットを使って、ラベル付けの間違いを見つけるブログもあるので、Food-101内のラベリングミスがあるのかもしれません(怪しいなと思う画像はいくつかありました)。
おわりに
甘雨が好きです。
パイモンはそこまで好きじゃないです。
Appendix
二クラス分類交差検証結果
3回の平均精度:0.99978246
- 1回目
- 精度:0.99978244
- 混同行列
[[2099 0] [ 1 2497]]
- 2回目
- 精度:0.99956495
- 混同行列
[[2099 0] [ 3 2495]]
- 3回目
- 精度:1.0
- 混同行列
[[2099 0] [ 0 2498]]
参考文献
ネットワークサービスにおける著作物の利用に関するガイドライン
Genshin Impact – Step Into a Vast Magical World of Adventure
Food-101 -- Mining Discriminative Components with Random Forests
GitHub - stratospark/food-101-keras: Food Classification with Deep Learning in Keras / Tensorflow
ラベルの付け間違いが分かる!?|京セラコミュニケーションシステム(KCCS)
Bossard, Lukas and Guillaumin, Matthieu and Van Gool, Luc, Food-101 – Mining Discriminative Components with Random Forests, European Conference on Computer Vision, 2014.
C. Szegedy, V. Vanhoucke, S. Ioffe, and J. Shlens. Rethinking the inception architecture for computer vision. In Proc. of CVPR, 2016.
機械学習(pcaとkmeans)による画像のグルーピング
はじめにのはじめに
本記事で出てくる画像は一部、以下から引用してます。
株式会社MIHOYO corp.mihoyo.co.jp
はじめに
自分で集めた画像でCNNするために、TwitterAPIの検索機能を使って画像を集めています。
集めている画像は特定の作品のキャラクターだったりするわけですが、CNNで分類モデルを作る上で画像にラベルを付けなくてはなりません。
このラベル付けは言うまでもなく面倒で、時間がかかります。そこである程度自動化できないかと思ったわけです。
したがって、今回はタイトルにある通り、pca(主成分分析)とkmeans(クラスタリング)を用いて画像のグルーピングを行いたいと思います。
データ
キャラクターの画像は顔画像だけにあらかじめトリミングしてあるものを用います。
その手法を知りたい方は以下を参照ください。
古い記事ですが、lbpcascade_animeface.xmlはGoogleで検索した限りかなり広く使われています。
検索すればいくらでも出てくるかと思いますので、オリジナルのものを今回は添付させていただきました。
- ブログ記事
問題
では、具体的に画像のラベル付けで何が面倒かを以下に挙げます。
キャラクターの顔認識精度に限界があり、誤検知が発生するため、顔画像以外の画像がデータ内に混在する
特定のキャラクター1人のみラベルをつける場合、1枚1枚人が判定するのは時間がかかる
Twitterから集めているデータのため、ほぼ同じ画像がいくつか集まってしまう
1番の問題
lbpcascade_animeface.xmlは非常に良くできていますが、さすがに完璧な検知精度はありません。
opencv側のパラメータをいじることで、ある程度は見落とさないよう検知したり過検知することは防げますが、それにも限界はあります。
そのため、顔画像ではない画像が一つのクラスタとしてグルーピングされれば非常に手間が省けます。
結論を先に述べると、残念ながら本記事ではこの問題は解決できておりませんのでご注意ください。
2番の問題
今回の場合、2021年2月現在、話題のゲーム「原神」のキャラクターの画像をTwitterから集めているわけですが、この中で筆者が欲しい画像はパイモンだけです。
なぜパイモンの画像ばかり集めているかは別の記事で書くとして、パイモン以外の画像はすべて不要ということになります。
クラスタリングをすることで、あるクラスタに一人もパイモンがいなければ、そのクラスタのディレクトリ名で保存されている画像は不必要な画像として扱えます。
特定のキャラクターの画像を集める場合、こうすることで非常に効率が良くなることがわかるかと思います。
3番の問題
TwitterAPIの検索機能を使って、例えば「パイモン 原神」のように画像を集めるわけですが、どうしても同じ画像ばかり収集してしまうことがあります。
これを問題視するかどうかはCNNの学習に関わるかと思いますが、削除したいモチベーションがあるなら、これもクラスタリングで解決できます。
あるクラスタに同じ画像が集まれば、その画像の内、一枚を除いて削除すれば画像の重複がなくなるからです。
ps
これらの問題はクラスタリングすることでラベル付けの手間を省くことに注力しているため、最終的には人の判断が介入します。
プログラム
全体
import glob as gb import shutil import cv2 import os from sklearn.cluster import KMeans from sklearn.decomposition import PCA from sklearn.preprocessing import StandardScaler import numpy as np import pandas as pd import matplotlib.pyplot as plt import matplotlib.ticker as ticker import seaborn as sns from mpl_toolkits.mplot3d import Axes3D # 画像の読み込み def load_image(path): image_list = [] npy_image_list = [] image_path_list = gb.glob(path) # 画像データを一枚ずつ読み込む for i, image in enumerate(image_path_list): image_path = image.replace(chr(92), '/') # \を/に置換(windows特有)->macはchr(165) if i % 100 == 0: # 雑に進行状況出力 print(i) img_npy = cv2.imread(image_path, cv2.IMREAD_COLOR) # デフォルトカラー読み込み img_npy = cv2.cvtColor(img_npy, cv2.COLOR_BGR2RGB) # RGB変換 img_npy = cv2.resize(img_npy, (64, 64)) # リサイズ64x64 # plt.imshow(img_npy) # plt.show() # break img_npy = img_npy.flatten() # 一次元化 npy_image_list.append(img_npy/255) # 0~1に正規化 return npy_image_list # kmeansのモデル構築 def build_kmeans(df, cluster_num): kmeans = KMeans(n_clusters=cluster_num, random_state=2021) kmeans.fit(df) return kmeans # 主成分分析のモデル構築 def build_pca(df): pca = PCA() pca.fit(df) return pca # 主成分分析の累積寄与率を可視化(この結果をもとに特徴ベクトルを決める) def plot_contribution_rate(pca): fig = plt.figure() plt.gca().get_xaxis().set_major_locator(ticker.MaxNLocator(integer=True)) plt.plot([0] + list(np.cumsum(pca.explained_variance_ratio_)), "-o") plt.xlabel("Number of principal components") plt.ylabel("Cumulative contribution rate") plt.grid() plt.show() # plt.savefig('../figure/pca_contribution_rate.png') # 主成分分析の第一主成分と第二主成分で散布図による可視化 def plot_scatter2d(df): fig = plt.figure() sns.scatterplot(data=df, x='PC1', y='PC2', hue='label', palette='bright', legend='full') plt.show() # plt.savefig('../figure/pca_scatter2d.png') # 主成分分析の第一主成分と第二主成分と第三主成分で散布図による可視化 def plot_scatter3d(df): # https://qiita.com/maskot1977/items/082557fcda78c4cdb41f fig = plt.figure() ax = Axes3D(fig) #軸にラベルを付けたいときは書く ax.set_xlabel("PC1") ax.set_ylabel("PC2") ax.set_zlabel("PC3") #.plotで描画 for label in df['label'].values: ax.plot(df.loc[df['label']==label, 'PC1'], df.loc[df['label']==label, 'PC2'], df.loc[df['label']==label, 'PC3'], alpha=0.8, marker=".", linestyle='None') plt.show() # plt.savefig('../figure/pca_scatter3d.png') # 結果をクラスタごとにディレクトリに保存 def make_cluster_dir(load_path, save_path, kmeans): # 保存先のディレクトリを空にして作成 shutil.rmtree(save_path) os.mkdir(save_path) # クラスタごとのディレクトリ作成 for i in range(kmeans.n_clusters): cluster_dir = save_path + "cluster{}".format(i) if os.path.exists(cluster_dir): shutil.rmtree(cluster_dir) os.makedirs(cluster_dir) # 各ディレクトリにコピー保存 image_path_list = gb.glob(load_path) for label, path in zip(kmeans.labels_, image_path_list): shutil.copyfile(path, save_path + 'cluster{}/{}'.format(label, os.path.basename(path))) print('クラスタごとにファイル作成完了') def main(): LOAD_PATH = 'D:/Illust/Paimon/interim/face_only/*' # 画像データの読込先 SAVE_PATH = 'D:/Illust/Paimon/interim/face_only_clustering/' # 画像データをクラスタリングした結果の保存先 CSV_PATH = 'D:/Illust/Paimon/interim/face_only_pca.csv' # 画像データを主成分分析した結果の保存先 try: # すでに画像データを主成分分析した結果のCSVファイルがあれば読み込む、なければexceptへ pca_df = pd.read_csv(CSV_PATH) except FileNotFoundError: # 画像読み込み npy_image_list = load_image(LOAD_PATH) df = pd.DataFrame(npy_image_list) print(df.shape) # 主成分分析の実行 pca = build_pca(df) pca_df = pd.DataFrame(pca.transform(df), columns=["PC{}".format(x + 1) for x in range(len(df))]) plot_contribution_rate(pca) # 累積寄与率可視化 pca_df.to_csv(CSV_PATH, index=False) # 保存 # kmeansによるクラスタリング train_df = pca_df.iloc[:, :1200] # 学習データ cluster_num = int(input('cluster_num >')) # クラスタ数を入力 kmeans = build_kmeans(train_df, cluster_num) # kmeansモデル構築 make_cluster_dir(LOAD_PATH, SAVE_PATH, kmeans) # クラスタリング結果からディレクトリ作成 # 可視化 pca_df['label'] = kmeans.labels_ plot_scatter2d(pca_df) # 二次元散布図 plot_scatter3d(pca_df) # 三次元散布図 if __name__ == "__main__": main()
補足説明
全体の流れ
mainの例外処理
main関数の例外処理はCSV_PATHにあらかじめファイルを用意しない限り、原則except側に入ります。
画像を1枚1枚読み込んで主成分分析するのには、そこそこ時間がかかります。
何度もこの処理をするのは効率が悪いため、主成分分析した結果を一度実行したら保存しているわけです。二回目以降に実行時には保存した結果を読みに行きます。
主成分分析した結果がクラスタリングするときの特徴ベクトルになります。
try: # すでに画像データを主成分分析した結果のCSVファイルがあれば読み込む、なければexceptへ pca_df = pd.read_csv(CSV_PATH) except FileNotFoundError: # 画像読み込み npy_image_list = load_image(LOAD_PATH) df = pd.DataFrame(npy_image_list) print(df.shape) # 主成分分析の実行 pca = build_pca(df) pca_df = pd.DataFrame(pca.transform(df), columns=["PC{}".format(x + 1) for x in range(len(df))]) plot_contribution_rate(pca) # 累積寄与率可視化 pca_df.to_csv(CSV_PATH, index=False) # 保存
train_dfの1200
今回の場合、主成分分析した結果は約5000x5000の行列です。行が各主成分(つまり第5000主成分まである)、列が画像の枚数です。 主成分分析は次元圧縮のアルゴリズムで、5000ある特徴ベクトルを減らして(必要な情報だけに圧縮して)クラスタリング精度向上のために用います。 つまり、下記のコードで意味する1200は5000を1200まで減らしたということになります。
train_df = pca_df.iloc[:, :1200] # 学習データ
1200にした理由は、主成分分析の累積寄与率というものを用います。plot_contribution_rate(pca)関数はそれを可視化したものになります。
縦軸が1に近づけば近づくほど、その主成分だけでデータを説明できていることになります。
1200で限りなく1に近いと判断し、今回は1200にしました。600でも、1800でも試す価値はあると思います(今回はやっていません)。
なお、あらかじめ指定した累積寄与率を閾値として設定しておき、その値を超えたときの数を与えてやることもできます。

kmeansのクラスタ数
クラスタの数は標準入力で指定します。これをいくつにするかは正直適当です。
今回は50にしました。
cluster_num = int(input('cluster_num >')) # クラスタ数を入力 kmeans = build_kmeans(train_df, cluster_num) # kmeansモデル構築
参考までにクラスタリングした可視化結果を載せます。クラスタ数が50だと見えにくいので、クラスタ数を10で可視化しています。
- 二次元散布図

- 三次元散布図

結果
ディレクトリ全体

各ディレクトリ
うまくいった例
2番の問題を解決
このクラスタ数のディレクトリは特に触る必要がなくなります。

左上4番目にだけ凝光様がぽつんといます。白髪のため紛れ込んでしまったのでしょう。

今度は金髪のキャラクターが集まっているクラスタがありました。主人公がいっぱいいますね。一部クレーや凝光様もいるのが見てとれると思います(原神わからない方ごめんなさい)。
なお、今回は髪色で同じクラスタになっているケースは他にも多くありました。

3番の問題を解決
ほぼ同じ画像のパイモンばかりしかいません。お好みで画像を削除できます。


うまくいっていない例
しいて共通点を挙げるなら全体的に暗い画像となっている点です。しかし、明るい画像も一部含まれてもいます。
このような場合、髪色のクラスタにグルーピングされず、結果として共通点のないクラスタになっていましまいた。
今回うまくいっていないクラスタの大半がこのような暗めの画像でした。RGBだけでなくHSVの特徴ベクトルも必要かもしれませんね。
パイモンがいなければディレクトリごと除去できますが、実は真ん中あたりにしれっといます。

さて、これは本当に共通点がないクラスタです。カラー画像や白黒画像が紛れ込んでます。
同じ画像は同じクラスタには入っているようですが、それくらいしか共通点が見つかりませんでした。

まとめ
今回はpcaとkmeansを用いて画像のグルーピングをしました。結果はそこそこ実用的ではありますが、まだまだ精度向上はできそうな気がします。
今後の課題としては
- 1番の問題を解決する
- 画像の明るさを踏まえた特徴ベクトルを作成する
などがありますね。
あとは別の次元圧縮アルゴリズムとクラスタリングアルゴリズムを使ってみると良いかもしれません。正直pcaとkmeansは王道ワンパターンなので(ただ王道を馬鹿にできないのもまた事実)。
なお、最適なクラスタ数については考えません。kmeansでは正直不毛です。
クラスタリングアルゴリズムではxmeans使うか階層型クラスタリングを使うか。
次元圧縮アルゴリズムではt-SNEを使うか。
といったところでしょうか。
参考文献
ネットワークサービスにおける著作物の利用に関するガイドライン
Genshin Impact – Step Into a Vast Magical World of Adventure
【機械学習】ブログのサムネ画像をクラスタリングしてみる! - 株式会社ライトコード
Python: seaborn を使った可視化を試してみる - CUBE SUGAR CONTAINER
matplotlib 3次元散布図 | Python学習講座
[Python入門]shutilモジュールによる高水準ファイル操作:Python入門(1/3 ページ) - @IT
Pythonでファイル・ディレクトリを削除するos.remove, shutil.rmtreeなど | note.nkmk.me
カテゴリ変数(質的データ)の前処理の違いまとめ
はじめに
一般的に機械学習においてカテゴリ変数は、前処理として数値化する必要があります。
本記事ではその前処理の方法と違いについてまとめです。
データの種類と意味
下図のように変数は4つの尺度に分けられます。
今回説明するのは名義尺度と順序尺度に関する前処理の方法です。

データ
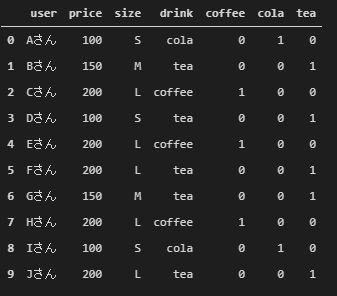
まず、簡単なデータを以下のコードで準備します。あるユーザがある飲み物を買ったときの値段、サイズ、飲み物の種類が記載されています。
このデータでは、sizeデータはL>M>Sの関係にあるため順序尺度、drinkデータは大小関係がないため名義尺度になります。
import numpy as np import pandas as pd df = pd.DataFrame([ ['Aさん', 100, 'S', 'cola'], ['Bさん', 150, 'M', 'tea'], ['Cさん', 200, 'L', 'coffee'], ['Dさん', 100, 'S', 'tea'], ['Eさん', 200, 'L', 'coffee'], ['Fさん', 200, 'L', 'tea'], ['Gさん', 150, 'M', 'tea'], ['Hさん', 200, 'L', 'coffee'], ['Iさん', 100, 'S', 'cola'], ['Jさん', 200, 'L', 'tea']], columns=['user', 'price', 'size', 'drink']) df

前処理の種類と違い
一般に2つのアプローチがあります。
1つ目は順番にラベリングしていく方法です。colaを0、teaを1、coffeeを2のようにラベルをつけます。
2つ目はダミー変数化(OneHotエンコーディング)する方法です。cola、tea、coffeeのカラムを新しく追加し、任意のカラムを1、それ以外のカラムを0にします。
1つ目の方法は決定木ベースのモデルには効果はありますが、線形モデルやNNに使う場合は注意が必要です。「teaはcoffeeと2倍の関係にある」といった解釈をされかねません。
2つ目の方法は1つ目より使われる気がします。ただし、カテゴリ変数が1000個あるといったふうに、量が多ければ多いほどカラムも増えます。膨大なカテゴリ変数は適宜集約するといった処理が必要になるかと思います。
名義尺度
ラベリング
factorize
pandasのfactorizeを使えば、簡単にカテゴリ変数をラベリングしてくれます。
ft_array, ft_index = pd.factorize(df['drink']) # tupple型で返却される df_ft = pd.DataFrame(ft_array, columns=['ft_drink']) df_factrize = pd.concat([df, df_ft], axis=1) # 元のデータフレームと連結 df_factrize

LabelEncoder
sklearnのLabelEncoderを使えば、簡単にカテゴリ変数をラベリングしてくれます。
from sklearn.preprocessing import LabelEncoder lenc = LabelEncoder() lenc.fit(df['drink']) lenc_vec = lenc.transform(df['drink']) df_le = pd.DataFrame(lenc_vec, columns=['le_drink']) df_lenc = pd.concat([df, df_le], axis=1) # 元のデータフレームと連結 df_lenc

factorizeとLabelEncoderの違い
以下のように学習データとテストデータに分けられたデータがあるとします。
Nさんが学習データにはないサイズと飲み物の種類(LLとcider)が記載されていることに注意してください。
train = df.copy()
test = pd.DataFrame([
['Kさん', 200, 'L', 'cola'],
['Lさん', 100, 'S', 'tea'],
['Mさん', 150, 'M', 'coffee'],
['Nさん', 250, 'LL', 'cider']],
columns=['user', 'price', 'size', 'drink'])
factorizeは値の出現順にラベルが振られるため、データフレームが別々にある場合、別のラベルが振らてしまう可能性があります。
train_ft, idx = pd.factorize(train['drink']) test_ft, idx = pd.factorize(test['drink']) train_df = pd.concat([train, pd.DataFrame(train_ft, columns=['ft_drink'])], axis=1) test_df = pd.concat([test, pd.DataFrame(test_ft, columns=['ft_drink'])], axis=1) display(train_df) display(test_df)

一度trainデータとtestデータを連結させ、1つのデータフレームとしてラベルを振れば回避することができます。
all_df = pd.concat([train, test], axis=0).reset_index(drop=True) all_ft, idx = pd.factorize(all_df['drink']) train_test_df = pd.concat([all_df, pd.DataFrame(all_ft, columns=['ft_drink'])], axis=1)
しかし、kaggleのようにあらかじめ学習データとテストデータがわけられている場合、わざわざ連結するのは面倒です。LabelEncoderを使えば連結せずに済みます。
なお、ラベリングの順番はfactorizeは値の出現順ですが、LabelEncoderはアルファベット順です。名義尺度のデータでは、ここはあまり気にする必要はないかと思います。
lenc = LabelEncoder() train_df = lenc.fit(['cola', 'tea', 'coffee', 'cider']) # ここでカテゴリ変数の種類を指定 train_lenc = lenc.transform(train[['drink']]) test_lenc = lenc.transform(test[['drink']]) train_df = pd.concat([train, pd.DataFrame(train_lenc, columns=['le_drink'])], axis=1) test_df = pd.concat([test, pd.DataFrame(test_lenc, columns=['le_drink'])], axis=1) display(train_df) display(test_df)

ダミー変数化
get_dummies
pandasのget_dummiesを使えば、簡単にカテゴリ変数をダミー変数化してくれます。
df_gd = pd.get_dummies(df['drink']) df_dummy = pd.concat([df, df_gd], axis=1) # 元のデータフレームと連結 df_dummy
OneHotEncoder
sklearnのOneHotEncoderを使えば、同様にダミー変数化してくれます。
from sklearn.preprocessing import OneHotEncoder oenc = OneHotEncoder(sparse=False, dtype=int) oenc.fit(df[['drink']]) # pandas.core.frame.DataFrame型もしくは二次元のnumpy.ndarray型が引数でないとエラー oenc_vec = oenc.transform(df[['drink']]) # numpy.ndarray型で返却される df_oenc = pd.DataFrame(oenc_vec, columns=['coffee', 'cola', 'tea']) df_oht = pd.concat([df, df_oenc], axis=1) # 元のデータフレームと連結 df_oht

LabelBinarizer
sklearnのLabelBinarizerを使えば、同様にダミー変数化してくれます。
from sklearn.preprocessing import LabelBinarizer lbnr = LabelBinarizer() lbnr.fit(df[['drink']]) df_lbnr = pd.concat([df, pd.DataFrame(lbnr.transform(df[['drink']]), columns=['coffee', 'cola', 'tea'])], axis=1) # OneHotEncoderとほぼ同じコードなためワンライナーで記述 df_lbnr

get_dummiesとOneHotEncoder、LabelBinarizerの違い
factorizeとLabelEncoderの違い同様、 学習データとテストデータにデータがわけられているとします。
get_dummisを用いた場合、学習データとテストデータで作成されるカラムの数が異なってしまいます。factorizeとLabelEncoderの違いと同じように学習データとテストデータを連結させる方法もありますが、OneHotEncoderとLabelBinarizerを使えば回避できます。
以下のコードではLabelBinarizerの例です。
lbnr = LabelBinarizer() lbnr.fit(train['drink']) display(pd.DataFrame(lbnr.transform(train[['drink']]), columns=['coffee', 'cola', 'tea'])) display(pd.DataFrame(lbnr.transform(test[['drink']]), columns=['coffee', 'cola', 'tea']))

ただし、データの中にnanもしくはinfが含まれている場合、get_dummisはエラーが出ませんが、OneHotEncoderとLabelBinarizerはエラーが出ます。
oenc = OneHotEncoder(sparse=False) df['nan_and_inf'] = ['A', 'A', 'A', 'A', 'A', 'A', np.nan, np.inf, 'B', 'B'] df

# エラーが出る oenc.fit_transform(df[['nan_and_inf']])
# エラーが出ない pd.get_dummies(df['nan_and_inf'])

nanはスルーされますが、infはされないことに注意です。
OneHotEncoderとLabelBinarizerの違い
OneHotEncoderとLabelBinarizerの違いは複数のカラムをまとめてダミー変数化できるかどうかです。順序尺度データではありますが、sizeデータもまとめてダミー変数化してみます。
# エラーが出る pd.DataFrame(lbnr.fit_transform(train[['drink', 'size']]), columns=['coffee', 'cola', 'tea', 'L', 'M', 'S'])
# エラーが出ない pd.DataFrame(oenc.fit_transform(train[['drink', 'size']]), columns=['coffee', 'cola', 'tea', 'L', 'M', 'S'])

順序尺度
名義尺度とは異なりラベリングが一般的かと思います。しかし、factorizeやLabelEncoderでは任意の順番にラベリングできません。また、色々調べてみましたが、pandas、sckit-learnともに任意の順番にラベリングできるメソッドはなさそうです。
そのため自分で関数なり作る必要があります。以下に例を示します。
apply
lambda式を使えば一行で書けます。
df['ordinal_size'] = df['size'].apply(lambda x: ['S', 'M', 'L', 'LL'].index(x))
map
mapでもlambda式を使えば一行で書けます。ただmapの場合は辞書型もいける口です。ここは個人の好みでしょう。
# df['ordinal_size'] = df['size'].map(lambda x: ['S', 'M', 'L', 'LL'].index(x)) # lambdaもいける df['ordinal_size'] = df['size'].map({'S': 0, 'M': 1, 'L': 2, 'LL':3})
また、自分でカテゴリ変数のリストを作るのが面倒な場合(もしくは多すぎる)は、学習データとテストデータ含めたすべてのデータからユニークな値を取るしかない思います。
unique_size_list = list(all_df['size'].unique()) train['ordinal_size'] = train['size'].apply(lambda x: unique_size_list.index(x)) test['ordinal_size'] = test['size'].apply(lambda x: unique_size_list.index(x)) display(train) display(test)

factorize
一応factorizeとLabelEncoderの実行結果も示します。
sizeの出現順がS、M、Lだったためたまたまうまくいっています。順番があらかじめラベリングしたい順序にソートされていればfactorizeは使えなくもないです。
ft_array, ft_index = pd.factorize(df['size']) # tupple型で返却される df_ft = pd.DataFrame(ft_array, columns=['ft_size']) df_factrize = pd.concat([df, df_ft], axis=1) # 元のデータフレームと連結 df_factrize

LabelEncoder
アルファベット順=ラベリングしたい順序であれば使えます。sizeの例では無理です。
from sklearn.preprocessing import LabelEncoder lenc = LabelEncoder() lenc.fit(df['size']) lenc_vec = lenc.transform(df['size']) df_le = pd.DataFrame(lenc_vec, columns=['le_size']) df_lenc = pd.concat([df, df_le], axis=1) # 元のデータフレームと連結 df_lenc

まとめ
まとめると以下の表になります。
- ○:この手法がベター、もしくはこの手法しかない
- △:この手法よりも良い手法がある
- ✕:この手法は一般的ではない
| 名義尺度 | 順序尺度 | 感想 | |
|---|---|---|---|
| get_dummis | △ | ✕ | 学習データとテストデータのカテゴリ変数に注意 |
| OneHotEncoder | ○ | ✕ | nanとinfに注意 |
| LabelBinarizer | ○ | ✕ | nanとinfに注意 |
| factorize | △ | △ | 楽だが中途半端 |
| LabelEncoder | △ | △ | 楽だが中途半端 |
| オリジナル関数 | △ | ○ | オレオレ関数は最強 |
表から分かる通り、名義尺度はOneHotEncoderもしくはLabelBinarizerを使う、順序尺度はオリジナル関数を定義する、というのが結論です。
参考文献
人工知能プログラミングのための数学がわかる本 | 石川 聡彦 |本 | 通販 | Amazon
Pythonでのカテゴリ変数(名義尺度・順序尺度)のエンコード(数値化)方法 ~順序のマッピング、LabelEncoderとOne Hot Encoder~ - Qiita
One-HotエンコーディングならPandasのget_dummiesを使おう | Shikoan's ML Blog
【python】機械学習でpandas.get_dummiesを使ってはいけない - 静かなる名辞
【python】sklearnでのカテゴリ変数の取り扱いまとめ LabelEncoder, OneHotEncoderなど - 静かなる名辞
pandasでカテゴリー変数を数値に変換する | 分析ノート
機械学習のおおまかな流れを理解する
この記事は SLP KBIT Advent Calendar 2020 12日目の記事です。(じゃあなぜ 13日に公開しているのか)
adventar.org
はじめに
機械学習を勉強する上で、重要なことはいくつもあると思います。
その中でも本記事は全体像の部分、機械学習のおおまかな流れに焦点を当てます。
機械学習を勉強し続けてはや2年。機械学習の分野は学ぶことが多く、勉強したてのときは気にも止めていなかったおおまかな流れですが、これを理解しているのとしていないとのでは今後の理解度に大きく響くと感じています。
ですので、本記事では〇〇モデルの具体的なアルゴリズムや数学的内容(理論)だったり、精度向上の前処理だったりは省いてますのでご了承を。
機械学習勉強したての後輩の誰かの目に入れば幸いです。
機械学習のおおまかな流れ
下図が機械学習のおおまかな流れです。大きく二つにわかれます。
一つ目は学習です。すでに手元にあるデータを学習データとして入力し、モデルを出力します。
モデルとはデータからある目的を解くために機械が分かる形に数値化(数式化)したものです。書いてて思いますが、非常にわかりにくい。
ある目的には大きく分類と回帰があります。分類は例えば犬と猫の画像を分類する。回帰は株価を予測するなどがあります。
二つ目は推論です。モデルと新しく入手したデータを現在データとしてマッチングさせることで、結果を出力します。
推論と書いてますが、これは分類や回帰など目的によって変わるため、一般的に推論と呼ばれています。そのため出力結果は二値分類なら0or1、回帰なら予測値になります。
長くなりましたが、この学習と推論がごっちゃにならないようにしようぜ、というのが言いたいことです。

線形回帰
機械学習を学ぶとき線形回帰から触れることが多いんじゃないでしょうか。数学的な理論を理解するには大学レベルの数学はいりますが、直感的なイメージは中学生でも理解できるからです。
Excelの散布図で近似曲線を引いたことがある人はすぐに理解できると思います。 そうです、y=ax+bです。
例えば人の身長を予測したいとします。身長が高ければ、体重も重くなるでしょう。これは正の相関があると言えます。正の相関があるということは体重からある程度身長がいくつか予測できることはなんとなくわかるかと思います。
ここで、身長(予測対象の変数)を一般的に目的変数と呼びます。対して体重(予測するために使う変数)を説明変数と言います。前者は一つ、後者は一つ以上と考えてください。
説明変数は今は体重だけです。人には痩せ型の人もいれば、太っている人もいるでしょう。そのため体重からだけで身長を正確に予測することは不可能に近いです。
では別に年齢、性別、手足の大きさ、腕の長さ、足の長さなどなど、人に関する情報がより詳しくデータとしてあるとしましょう。体重だけよりも正確に予測できることは想像できると思います。
線形回帰の話はこれくらいにしますが、より詳しく知りたい人は参考文献を参照してみてください。
データ
ボストン市郊外の地域別住宅価格のデータを使わさせていただきます。線形回帰を使う機械学習のデータセットとして一番有名だと思います。
プログラム
Pythonは機械学習関連のライブラリが豊富ですが、ライブラリがあまりにできすぎていて、ほとんど数行足らずで書き終わります。そのため実装は簡単でも本質的に理解しにくく、非常に混乱してしまいます。
その結果、世の中にある機械学習のコードはほとんど、学習と推論を一括で書かれているケースが多いです。数行で書けるゆえの弊害ですかね。
とは言ったものの、世の中の実際のデータは機械学習のデータセットのようにきれい整形されていないため、地獄の前処理に苦しむわけですが、ここでは割愛。
学習と推論を強調するためにモデルをファイルとして一度保存してます。
ライブラリを使い倒してますが、とりあえずそこは一旦横に置いておいて。
図と照らし合わせながら読むと良いかと思います。おおまかな流れだけを意識してみてください。
- X_train: 学習データ(説明変数)
- y_train: 学習データ(目的変数)
- X_test: 現在データ(説明変数)
- y_test: 現在データ(目的変数)
gistb11b42b7ac823d54430f5cacf418e4d2
まとめ
機械学習のおおまかな流れについてまとめました。
繰り返しになりますが学習と推論です。可視化などしてますが、説明変数を決めるためにしているだけです。
線形回帰の精度評価は本記事では行いません。当たり前ですが、何の前処理もしていないので、精度は低いことは明らかです。
ボストン市郊外の地域別住宅価格を扱った記事は多くあると思いますので、そちらをご参照を。
キーワードとしては標準化、重回帰あたりを調べると精度が向上するかと思います。
参考文献
http://archive.ics.uci.edu/ml/index.php https://qiita.com/0NE_shoT_/items/08376b08783cd554b02e https://momonoki2017.blogspot.com/2018/01/scikit-learn_28.html https://localab.jp/blog/save-and-load-machine-learning-models-in-python-with-scikit-learn/
画像が見えにくいところもあるので、GitHubのリンクも載せておきます。
オートエンコーダによる時系列データの教師なし異常検知
はじめに
深層学習を用いた異常検知手法では有名なオートエンコーダを使ってプログラミングしたことをまとめます。オートエンコーダによる再構成誤差とLSTMによる予測誤差などとも比較予定です。
対象データ
今回もこちらの心電図のデータを使わせていただきました。
心電図のデータですので比較的周期性のあるデータですね。では実装していきます。
なお、データに関しては以前異常検知に関するブログでまとめてあるのでご参照ください。 noleff.hatenablog.com
プログラム
環境
- OS: macOS Catalina
- numpy: 1.18.5
- pandas: 1.0.5
- matplotlib: 3.3.0
- scikit-learn: 0.23.1
- tensorflow: 2.3.0
- Keras: 2.4.3
関数
関数定義します。意味はコメントアウトを見てください。
# センサデータ取得 def read_df(path): df = pd.read_csv(path) return df # Timeから時間の差を追加 def differ_time(df): # Timeから時間の差を割り当てる df['dif_sec'] = df['time'].diff().fillna(0) df['cum_sec'] = df['dif_sec'].cumsum() return df # 正規化 def act_minxmax_scaler(df): scaler = MinMaxScaler() scaler.fit(df) # 正規化したデータを新規のデータフレームに # df_mc = pd.DataFrame(scaler.transform(df), columns=df.columns) # 正規化したデータをnumpyリストに mc_list = scaler.fit_transform(df) return mc_list # 部分時系列の作成 def split_part_recurrent_data(data_list, window): data_vec = [] for i in range(len(data_list)-window+1): data_vec.append(data_list[i:i+window]) return data_vec # オートエンコーダの層を作成 def create_auto_encorder(in_out_shape): model = Sequential() # エンコード model.add(Dense(units=200, activation='relu', input_shape=(in_out_shape,))) model.add(Dense(units=100, activation='relu')) model.add(Dense(units=50, activation='relu')) # デコード model.add(Dense(units=100, activation='relu')) model.add(Dense(units=200, activation='relu')) # 出力層 model.add(Dense(in_out_shape, activation='sigmoid')) # 作成したネットワークの確認 model.summary() return model # オートエンコーダ実行 def act_auto_encoder(model, train_vec, batch_size, epochs): # 学習条件の設定 誤差関数=平均二乗誤差、最適化手法=Adam法 model.compile(loss='mse', optimizer='adam', metrics=['acc']) # 学習の実行 hist = model.fit(x=train_vec, y=train_vec, batch_size=batch_size, verbose=1, epochs=epochs, validation_split=0.2) return hist # 評価系のグラフをプロット def plot_evaluation(eval_dict, col1, col2, xlabel, ylabel): plt.plot(eval_dict[col1], label=col1) plt.plot(eval_dict[col2], label=col2) plt.ylabel(ylabel) plt.xlabel(xlabel) plt.legend() plt.show()
以下ベタ書き
前処理
まずデータを読み込んで、学習データとテストデータにわけます。
# データ読み込み df = read_df('./ecg.csv') # 使うデータだけ抽出 test_df = df.iloc[:5000, :] train_df = df.iloc[5000:, :]
グラフにするとこんな感じ。
plt.figure(figsize=(16,4)) plt.plot(train_df['signal2'], label='Train') plt.plot(test_df['signal2'], label='Test') plt.legend() plt.show()

秒数を計算して、時系列情報を秒として持ちます。 差分をとったdif_secを加算して、cum_secカラムを新規追加します。
# 秒数を計算
test_df = differ_time(test_df)
train_df = differ_time(train_df)
display(test_df)
display(train_df)

教師なしで異常検知する上で最終的に評価したいので正解のラベルを振ります。
今回は完全にグラフを目で見て、勝手に決めました。
そのため、これが実際に異常かどうかはわかりません。注意してください。
テストデータのグラフを15秒から20秒の間でクローズアップしました。
赤色に示すとおり、17秒から17.5秒の間を異常としました。
anomaly_df = test_df[(test_df['cum_sec']>15)&(test_df['cum_sec']<20)] plt.figure(figsize=(16,4)) plt.plot(anomaly_df['cum_sec'], anomaly_df['signal2'], color='tab:orange', label='Test') anomaly_df = test_df[(test_df['cum_sec']>17)&(test_df['cum_sec']<17.5)] plt.plot(anomaly_df['cum_sec'], anomaly_df['signal2'], color='tab:red', label='Test') plt.xlabel('sec') plt.show()

赤色の部分にラベル1、それ以外をラベル0にして、新規カラムlabelを定義します。
test_df.loc[(test_df['cum_sec']>17)&(test_df['cum_sec']<17.5), 'label'] = 1 test_df['label'] = test_df['label'].fillna(0)
次に部分時系列を作ってきます。 異常検知には「外れ値検知」「変化点検知」「異常部位検知」の大きく3種類ががあります。 3種類の異常に関してはこちらを参照ください。
今回の心電図データは「異常部位検知」に該当します。
異常部位検知において、言葉の通り、ある時系列の部位を異常として検知したいです。そこで時系列を部分ごとに分割した部分時系列を作ります。
生データそのままだとうまく学習できないため、正規化してデータを0~1の範囲に値を収めます。
window = 250 # 部分窓 # 正規化 train_vec = act_minxmax_scaler(train_df[['signal2']]) test_vec = act_minxmax_scaler(test_df[['signal2']]) # 一次元リスト train_vec = train_vec.flatten() test_vec = test_vec.flatten() # 部分時系列作成 train_vec = split_part_recurrent_data(train_vec, window) test_vec = split_part_recurrent_data(test_vec, window)
学習
続いてメインのオートエンコーダです。学習するための層を構築していきます。
in_out_shapeはオートエンコーダの入力と出力のデータ数です。今回の場合はwindowと同じです。
オートエンコーダの中間層は適当に決めています。ここをチューニングすることこそが、ニューラルネットワークの醍醐味といえるでしょう。
in_out_shape = window
# オートエンコーダ作成
model = create_auto_encorder(in_out_shape)
Model: "sequential" _________________________________________________________________ Layer (type) Output Shape Param # ================================================================= dense (Dense) (None, 200) 50200 _________________________________________________________________ dense_1 (Dense) (None, 100) 20100 _________________________________________________________________ dense_2 (Dense) (None, 50) 5050 _________________________________________________________________ dense_3 (Dense) (None, 100) 5100 _________________________________________________________________ dense_4 (Dense) (None, 200) 20200 _________________________________________________________________ dense_5 (Dense) (None, 250) 50250 ================================================================= Total params: 150,900 Trainable params: 150,900 Non-trainable params: 0 _________________________________________________________________
学習を実行させていきます。オートエンコーダは学習データとして入力したデータを再現するように学習するニューラルネットワークです。
そのため、説明変数と目的変数は同じtrain_vecが入ります(act_auto_encoder関数の話)。
batch_size = 100 epochs = 20 train_vec = np.array(train_vec) test_vec = np.array(test_vec) hist = act_auto_encoder(model, train_vec, batch_size, epochs)
Epoch 1/20 318/318 [==============================] - 1s 3ms/step - loss: 0.0090 - acc: 0.0584 - val_loss: 0.0014 - val_acc: 0.1011 Epoch 2/20 318/318 [==============================] - 1s 3ms/step - loss: 0.0011 - acc: 0.1096 - val_loss: 7.4762e-04 - val_acc: 0.0962 Epoch 3/20 318/318 [==============================] - 1s 3ms/step - loss: 6.4285e-04 - acc: 0.1120 - val_loss: 5.4386e-04 - val_acc: 0.0996 Epoch 4/20 318/318 [==============================] - 1s 3ms/step - loss: 5.2786e-04 - acc: 0.1153 - val_loss: 4.9082e-04 - val_acc: 0.1028 Epoch 5/20 318/318 [==============================] - 1s 3ms/step - loss: 4.7851e-04 - acc: 0.1195 - val_loss: 4.2493e-04 - val_acc: 0.1082 Epoch 6/20 318/318 [==============================] - 1s 3ms/step - loss: 4.3090e-04 - acc: 0.1236 - val_loss: 3.7944e-04 - val_acc: 0.1099 Epoch 7/20 318/318 [==============================] - 1s 3ms/step - loss: 4.0277e-04 - acc: 0.1275 - val_loss: 3.7550e-04 - val_acc: 0.1146 Epoch 8/20 318/318 [==============================] - 1s 3ms/step - loss: 3.8248e-04 - acc: 0.1302 - val_loss: 3.4081e-04 - val_acc: 0.1204 Epoch 9/20 318/318 [==============================] - 1s 3ms/step - loss: 3.7038e-04 - acc: 0.1336 - val_loss: 3.3807e-04 - val_acc: 0.1229 Epoch 10/20 318/318 [==============================] - 1s 3ms/step - loss: 3.5362e-04 - acc: 0.1362 - val_loss: 3.4684e-04 - val_acc: 0.1269 Epoch 11/20 318/318 [==============================] - 1s 3ms/step - loss: 3.4186e-04 - acc: 0.1391 - val_loss: 3.1406e-04 - val_acc: 0.1294 Epoch 12/20 318/318 [==============================] - 1s 3ms/step - loss: 3.2798e-04 - acc: 0.1446 - val_loss: 3.1423e-04 - val_acc: 0.1332 Epoch 13/20 318/318 [==============================] - 1s 3ms/step - loss: 3.2826e-04 - acc: 0.1473 - val_loss: 2.9715e-04 - val_acc: 0.1377 Epoch 14/20 318/318 [==============================] - 1s 3ms/step - loss: 3.1464e-04 - acc: 0.1475 - val_loss: 3.0181e-04 - val_acc: 0.1439 Epoch 15/20 318/318 [==============================] - 1s 3ms/step - loss: 3.0473e-04 - acc: 0.1517 - val_loss: 2.9136e-04 - val_acc: 0.1446 Epoch 16/20 318/318 [==============================] - 1s 4ms/step - loss: 2.9575e-04 - acc: 0.1564 - val_loss: 2.9008e-04 - val_acc: 0.1465 Epoch 17/20 318/318 [==============================] - 1s 3ms/step - loss: 2.9276e-04 - acc: 0.1569 - val_loss: 2.8658e-04 - val_acc: 0.1494 Epoch 18/20 318/318 [==============================] - 1s 3ms/step - loss: 2.8440e-04 - acc: 0.1574 - val_loss: 2.7905e-04 - val_acc: 0.1521 Epoch 19/20 318/318 [==============================] - 1s 3ms/step - loss: 2.7920e-04 - acc: 0.1611 - val_loss: 2.6296e-04 - val_acc: 0.1561 Epoch 20/20 318/318 [==============================] - 1s 4ms/step - loss: 2.7432e-04 - acc: 0.1648 - val_loss: 2.5073e-04 - val_acc: 0.1508
戻り値histから、誤差値と評価値の収束具合を描画します。
val_lossよりもlossが大きければ未学習の可能性が高いです。
# 誤差の収束具合を描画 plot_evaluation(hist.history, 'loss', 'val_loss', 'epoch', 'loss')

val_accよりもaccが大きければ過学習の可能性が高いです。
# 評価の収束具合を描画 plot_evaluation(hist.history, 'acc', 'val_acc', 'epoch', 'acc')

異常検知
作成したモデルから予測してみます。
異常部位の予測が変になってますね。
# モデルをテストデータに適用 pred_vec = model.predict(test_vec) # テストデータと出力データの可視化 plt.plot(test_vec[:,0], label='test') plt.plot(pred_vec[:,0], alpha=0.5, label='pred') plt.legend() plt.show()

テストデータとテストデータを再現したデータの再構成誤差を計算します。シンプルな距離の計算です。
グラフをみると、まあまあうまくできているように見えます。
# 再構成誤差(異常スコア)の計算 dist = test_vec[:,0] - pred_vec[:,0] dist = pow(dist, 2) dist = dist / np.max(dist) # 異常スコアの可視化 plt.plot(dist) plt.show()

もとのデータと比べてみるとこんな感じです。
plt.plot(test_vec[3800:,0], label='test') plt.plot(pred_vec[3800:,0], label='pred') plt.plot(dist[3800:], label='dist') plt.legend() plt.ylabel('anomaly score') plt.show()

評価
実際に評価してみます。 まず、異常度からラベルが0か1かを決めます。異常度は0から1に収まりますので、試しに0.8以上を異常としてみます。
pred_label_list = [1 if d >= 0.8 else 0 for d in dist] pred_df = pd.DataFrame(pred_label_list, columns=['pred_label']) new_test_df = pd.concat([test_df, pred_df], axis=1) new_test_df = new_test_df.fillna(0)
0と1の離散型のデータをグラフにしているため、カクカクしてます。
青色が初めに定義した異常のラベル。オレンジ色が異常度が0.8以上を異常としたラベルです。
plt.plot(new_test_df['label'], label='true anomaly') plt.plot(new_test_df['pred_label'], label='pred anomaly') plt.ylabel('anomary label') plt.legend()

分類タスクで評価に使われる混同行列から、精度、適合率、再現率を計算します。
print(confusion_matrix(new_test_df['label'], new_test_df['pred_label'])) print('accuracy_score: ', accuracy_score(new_test_df['label'], new_test_df['pred_label'])) print('precision_score: ', precision_score(new_test_df['label'],new_test_df['pred_label'])) print('recall_score: ', recall_score(new_test_df['label'],new_test_df['pred_label']))
果たしてこれはうまくいっていると言えるでしょうか。
結果から分かる通り、異常の誤検知は一回もしていません。それは適合率から明らかです。
ですが、再現率が非常に低いことから、多くの異常を見逃していることがわかります。
[[4876 0] [ 110 14]] accuracy_score: 0.978 precision_score: 1.0 recall_score: 0.11290322580645161
ここで異常度からラベルを振る閾値をまじめに考えてみます。
まずは異常度をヒストグラムで描画してみます。
ほとんど0近辺の値しかないことがわかります。
つまり、閾値0.8は本当にかけ離れた異常度のみを異常としてラベルをふっていることになります。

閾値を0.1 以上にしてみましょう。
閾値を下げたのでもちろん誤検知の数は増えます。しかし、同時に異常の見逃しの数を減らすことができました。
誤検知と見逃しはトレードオフの関係なので、どういう異常検知をしたいかで閾値を調整する必要があります。
pred_label_list = [1 if d >= 0.1 else 0 for d in dist] pred_df = pd.DataFrame(pred_label_list, columns=['pred_label']) new_test_df = pd.concat([test_df, pred_df], axis=1) new_test_df = new_test_df.fillna(0) plt.plot(new_test_df['label'], label='true anomaly') plt.plot(new_test_df['pred_label'], label='pred anomaly') plt.ylabel('anomary label') plt.legend() plt.savefig('figure/11_0.1以上の異常ラベル.png') print(confusion_matrix(new_test_df['label'], new_test_df['pred_label'])) print('accuracy_score: ', accuracy_score(new_test_df['label'], new_test_df['pred_label'])) print('precision_score: ', precision_score(new_test_df['label'],new_test_df['pred_label'])) print('recall_score: ', recall_score(new_test_df['label'],new_test_df['pred_label']))
[[4852 24] [ 46 78]] accuracy_score: 0.986 precision_score: 0.7647058823529411 recall_score: 0.6290322580645161

まとめ
オートエンコーダによる時系列データの教師なし異常検知をプログラミングしました。
時系列データは有名な心電図のデータです。
簡単にプログラムしたため、異常検知の精度はいまいちです。心電図の生データを正規化しただけの前処理しかしていないため、もう少し工夫をする必要があるかもしれません。
また、オートエンコーダの層の次元数に関しても色々試してみる価値はあると思います。
参考文献
https://www.renom.jp/ja/notebooks/tutorial/time_series/lstm-ae-ad/notebook.html
https://note.nkmk.me/python-tensorflow-keras-basics
https://engineer-fumi.hatenablog.com/entry/2018/09/18/161914
LightGBMを使って気温予測してみた
はじめに
今回の記事では、Kagglerランカー達がこぞって使ってるという「LightGBM」なるものを使ってプログラミングしたことをまとめていきます。
LightGBMとは
LightGBMとは決定木アルゴリズムを応用したアルゴリズムです。よくXGBoostと比較されるのを目にします。
詳しくは以下のサイトをご参考ください。決定木、アンサンブル学習、勾配ブースティング(LightGBMやXGBoost)の順に、非常にわかりやすくまとめられています。
プログラミング
データ
実際に手を動かしていきます。まず、肝心のデータですが、今回は高松市の気象データを気象庁の方からダウンロードさせていただきました。
気象データの期間は2019-01-01から2020-08-29までです。
目標
今回の目標は「一時間単位に高松市の気温を予測する」こととします。そのためダウンロードした気象データは一日単位ではなく、一時間単位の気象データを収集しました。
プログラム
先に関数を定義しておきます。
# データ読み込み def read_df(path): df = pd.read_csv(path, sep=',') return df # データ抽出 def extract_train_test_data(df): train_df = df[df['year'] == 2019] test_df = df[(df['year'] == 2020)] return train_df, test_df # 学習、テストデータ作成 def make_train_test_data(df, col_list): train_df, test_df = extract_train_test_data(df) # 学習用、テスト用にデータを分割 # X_train = train_df[col_list] X_train = train_df.drop(['datetime', 'temperature'], axis=1) y_train = train_df['temperature'] # X_test = test_df[col_list] X_test = test_df.drop(['datetime', 'temperature'], axis=1) y_test = test_df['temperature'] return X_train, y_train, X_test, y_test
まず、データを読み込んで表示します。read_df関数の引数のファイル名は任意です。
# データ読み込み df = read_df('20190101-20200829-takamatsu.csv') df
気象庁のデータをそのまま使うと、pandasを使う上で不都合が多いので、以下のように整形しています。約14500レコードになりました。

カラムそれぞれの意味は以下の表にまとめます。
| カラム名 | 意味 |
|---|---|
| datetime | 日付 |
| temperature | 気温 |
| pressure | 気圧 |
| relative_humidity | 相対湿度 |
| wind_speed | 風速 |
| rainfall | 降水量 |
| sea_lavel_pressure | 海面気圧 |
| day_length | 日照時間 |
| year | 年 |
| month | 月 |
| day | 日 |
次に、気象データを学習用とテスト用に分割します。今回は2019の気象データを学習データ、2020の気象データをテストデータとしました。
col_listは目的変数である気温を予測するために使用する説明変数のカラムです。今回は使用していません。 特徴量選択や特徴量エンジニアリングについて触れず、気温と日付以外のすべてのカラムを説明変数(特徴量)とします。
# 学習、検証データ作成 col_list = ['relative_humidity', 'pressure', 'day_length', 'month', 'day', 'hour'] X_train, y_train, X_test, y_test = make_train_test_data(df, col_list)
LightGBM用にデータをセットし、学習データで回帰モデルを作成、テストデータで予測をします。
実際の2020-01-01から2020-08-29の気温データと、予測した結果を連結し、predicted_dfにまとめます。
評価基準はrmse(Root Mean Square Error)としています。
# LightGBM用のデータセット lgb_train = lgb.Dataset(X_train, y_train) lgb.test = lgb.Dataset(X_test, y_test) # 評価基準 params = {'metric' : 'rmse'} # 回帰モデル作成 gbm = lgb.train(params, lgb_train) # 予測 test_predicted = gbm.predict(X_test) predicted_df = pd.concat([y_test.reset_index(drop=True), pd.Series(test_predicted)], axis = 1) predicted_df.columns = ['true', 'pred'] predicted_df

予測値と実測値の散布図を作成してみます。散布図と一緒に赤色で y = x の一次関数も描画しました。赤色にデータが近いほど良い結果が出ていると言えます。
# 予測値と実測値の散布図 RMSE = np.sqrt(mean_squared_error(predicted_df['true'], predicted_df['pred'])) plt.figure(figsize = (4,3)) plt.scatter(predicted_df['true'], predicted_df['pred']) plt.plot(np.linspace(0, 40), np.linspace(0, 40), 'r-') plt.xlabel('True Temperature') plt.ylabel('Predicted Temperature') plt.text(0.1, 30, 'RMSE = {}'.format(str(round(RMSE,3))), fontsize=10) plt.show()

今度は予測値と実測値をそれぞれ折れ線図として作成してみます。横軸は2020-01-01 01:00:00 からの経過時間(一時間単位)です。
# 予測値と実測値の折れ線図 plt.figure(figsize=(16,4)) plt.plot(predicted_df['true'], label='true') plt.plot(predicted_df['pred'], label='pred') plt.xlabel('time') plt.ylabel('temperature') # plt.xlim(0,1000) plt.legend() plt.show()

大体うまくいけているのではないでしょうか。
もう少しクローズアップしてデータを見てみます。上記のプログラムにplt.xlimメソッドを使って横軸に制限をかけたときの折れ線図を示します。
0-1000の図で、横軸が500から600にかけてのところで予測値と実測値がずれていることがわかります。
- 0-100

- 0-1000

最後に有効な特徴量が何だったのか見てみます。
dayやmonthなどが上位に来ています。
# 特徴量の重要度 lgb.plot_importance(gbm, height = 0.5, figsize = (4,8)) plt.show()

所感
思ったよりもうまくできたので正直驚きました。Lightと名前につくだけあってとても高速です。
ハイパーパラメータをチューニングしたり、特徴量を選択してないため実装も簡単です。
検討すれば精度はさらに向上すると思います。
参考文献
Kaggler がよく使う「LightGBM」とは?【機械学習】 – 株式会社ライトコード
LightGBM 徹底入門 – LightGBMの使い方や仕組み、XGBoostとの違いについて
【機械学習実践】LightGBMで回帰してみる【Python】
【データサイエンティストへの道⑴】lightGBMの軽ーい実装とまとめ|すりふと/医学生|note
Appendix
今回のデータのペアプロットと相関係数を以下に添付します。
このように可視化することで、目的変数との相関を見比べれば、特徴量選択を決める一つの指標になると思います。
- ペアプロット

- 相関
