LSTMによる時系列データの教師なし異常検知
はじめに
以前(といってもかなり前)、オートエンコーダによる時系列データの教師なし異常検知に関して記事を書きました。
今回はその続きで、同じ心電図データを用いて、LSTMによる異常検知をしたいと思います。
プログラム
import numpy as np import pandas as pd import matplotlib.pyplot as plt from sklearn.preprocessing import MinMaxScaler from tensorflow.keras.models import Sequential from tensorflow.keras.layers import Dense from sklearn.model_selection import train_test_split from tensorflow.keras.callbacks import EarlyStopping from tensorflow.keras.models import Sequential from tensorflow.keras.layers import Dense, LSTM # Timeから時間の差を追加 def differ_time(df): # Timeから時間の差を割り当てる df['dif_sec'] = df['time'].diff().fillna(0) df['cum_sec'] = df['dif_sec'].cumsum() return df # 正規化 def act_minxmax_scaler(df): scaler = MinMaxScaler() scaler.fit(df) # 正規化したデータをnumpyリストに mc_list = scaler.transform(df) return mc_list, scaler # データ準備 def split_part_recurrent_data(data_list, look_back): X, Y = [], [] for i in range(len(data_list)-look_back-1): X.append(data_list[i:(i+look_back), 0]) Y.append(data_list[i + look_back, 0]) return np.array(X), np.array(Y) def create_lstm(look_back): model = Sequential() model.add(LSTM(4, input_shape=(look_back, 1))) model.add(Dense(1)) model.compile(loss='mean_squared_error', optimizer='adam') return model # LSTM学習 def act_lstm(model, train_x, train_y, batch_size, epochs): train_x, val_x, train_y, val_y = train_test_split(train_x, train_y, test_size=0.25, shuffle=False, random_state=2021) early_stopping = EarlyStopping(monitor='val_loss', min_delta=1.0e-3, patience=20, verbose=1) history = model.fit(train_x, train_y, epochs=epochs, batch_size=batch_size, verbose=2, validation_data=(val_x, val_y), callbacks=[early_stopping] ) return model, history
データの読み込みから、可視化までは、オートエンコーダのときと同じです。
df = pd.read_csv('ecg.csv') # 使うデータだけ抽出 test_df = df.iloc[:5000, :] train_df = df.iloc[5000:, :] # 時間カラム追加 train_df = differ_time(train_df) test_df = differ_time(test_df)
plt.figure(figsize=(16,4)) plt.plot(train_df['signal2'], label='Train') plt.plot(test_df['signal2'], label='Test') plt.legend() plt.show()

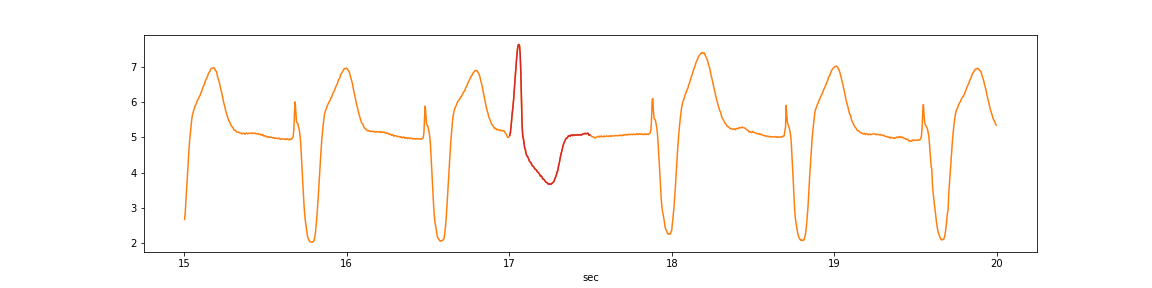
異常の該当箇所は赤色の場所と仮定します。
anomaly_df = test_df[(test_df['cum_sec']>15)&(test_df['cum_sec']<20)] plt.figure(figsize=(16,4)) plt.plot(anomaly_df['cum_sec'], anomaly_df['signal2'], color='tab:orange', label='Test') anomaly_df = test_df[(test_df['cum_sec']>17)&(test_df['cum_sec']<17.5)] plt.plot(anomaly_df['cum_sec'], anomaly_df['signal2'], color='tab:red', label='Test') plt.xlabel('sec') plt.show()
データ準備として正規化し、部分時系列にしたデータを用意します。
LSTMのデータ準備の詳細はこちらをご参考ください。
なお、look_backが250なのは、オートエンコーダの記事と合わせているだけです。
# 正規化 train_vec, train_scaler = act_minxmax_scaler(train_df[['signal2']]) test_vec, test_scaler = act_minxmax_scaler(test_df[['signal2']]) look_back = 250 train_x, train_y = split_part_recurrent_data(train_vec, look_back) test_x, test_y = split_part_recurrent_data(test_vec, look_back) display(pd.DataFrame(train_x)) display(pd.DataFrame(train_y))
今回、LSTMのライブラリはKerasを用います。
そのため、Kerasのお作法として、入力データを三次元にしなければりません。
そのためreshape処理をします。
# %% print(train_x.shape) print(test_x.shape) # [データ数, 部分時系列数(look_back), 特徴量数(心電の波形のみ)]へ変形 train_x = np.reshape(train_x, (train_x.shape[0], train_x.shape[1], 1)) test_x = np.reshape(test_x, (test_x.shape[0], test_x.shape[1], 1)) print(train_x.shape) print(test_x.shape) #(39749, 250) #(4749, 250) #(39749, 250, 1) #(4749, 250, 1)
# モデル作成 model = create_lstm(look_back) batch_size = 100 epochs = 100 # モデル学習 model, hist = act_lstm(model, train_x, train_y, batch_size, epochs)
Train on 29811 samples, validate on 9938 samples Epoch 1/100 29811/29811 - 5s - loss: 0.0066 - val_loss: 0.0024 Epoch 2/100 29811/29811 - 3s - loss: 0.0014 - val_loss: 8.8507e-04 Epoch 3/100 29811/29811 - 3s - loss: 8.1925e-04 - val_loss: 7.0201e-04 Epoch 4/100 29811/29811 - 3s - loss: 6.6374e-04 - val_loss: 5.8554e-04 Epoch 5/100 29811/29811 - 3s - loss: 5.7173e-04 - val_loss: 5.2490e-04 Epoch 6/100 29811/29811 - 3s - loss: 5.0133e-04 - val_loss: 4.8002e-04 Epoch 7/100 29811/29811 - 3s - loss: 4.4123e-04 - val_loss: 3.9693e-04 Epoch 8/100 29811/29811 - 3s - loss: 3.9236e-04 - val_loss: 3.5257e-04 Epoch 9/100 29811/29811 - 3s - loss: 3.5195e-04 - val_loss: 3.1569e-04 Epoch 10/100 29811/29811 - 3s - loss: 3.1859e-04 - val_loss: 2.9057e-04 Epoch 11/100 29811/29811 - 3s - loss: 2.9321e-04 - val_loss: 2.6281e-04 Epoch 12/100 29811/29811 - 3s - loss: 2.7145e-04 - val_loss: 2.4727e-04 Epoch 13/100 29811/29811 - 3s - loss: 2.5357e-04 - val_loss: 2.2870e-04 Epoch 14/100 29811/29811 - 3s - loss: 2.4035e-04 - val_loss: 2.1622e-04 Epoch 15/100 29811/29811 - 3s - loss: 2.2892e-04 - val_loss: 2.1792e-04 Epoch 16/100 29811/29811 - 3s - loss: 2.2230e-04 - val_loss: 1.9697e-04 Epoch 17/100 29811/29811 - 3s - loss: 2.1219e-04 - val_loss: 1.9251e-04 Epoch 18/100 29811/29811 - 3s - loss: 2.0502e-04 - val_loss: 1.8317e-04 Epoch 19/100 29811/29811 - 2s - loss: 1.9737e-04 - val_loss: 1.7637e-04 Epoch 20/100 29811/29811 - 3s - loss: 1.8952e-04 - val_loss: 1.7019e-04 Epoch 21/100 29811/29811 - 3s - loss: 1.8515e-04 - val_loss: 1.6518e-04 Epoch 22/100 29811/29811 - 2s - loss: 1.7755e-04 - val_loss: 1.7475e-04 Epoch 00022: early stopping
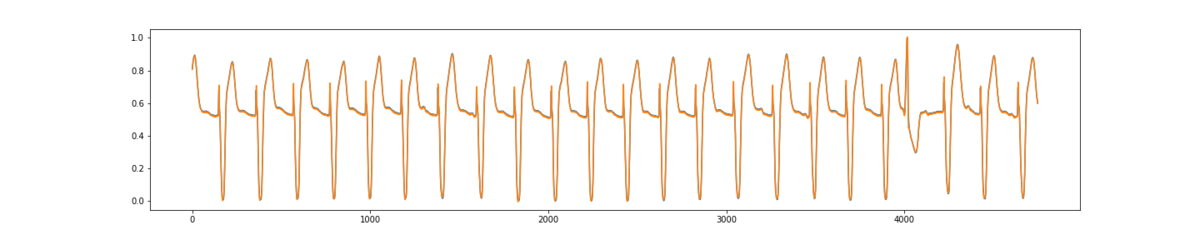
予測値と実測値を比較します。
異常箇所含め、かなり予測できてるかと思います。
# 予測 test_pred = model.predict(test_x) plt.figure(figsize=(20,4)) plt.plot(test_y) plt.plot(test_pred) plt.show()
ただ、精度良く予測することではなく、異常を見つけることなので、予測値と実測値の誤差を求めます。
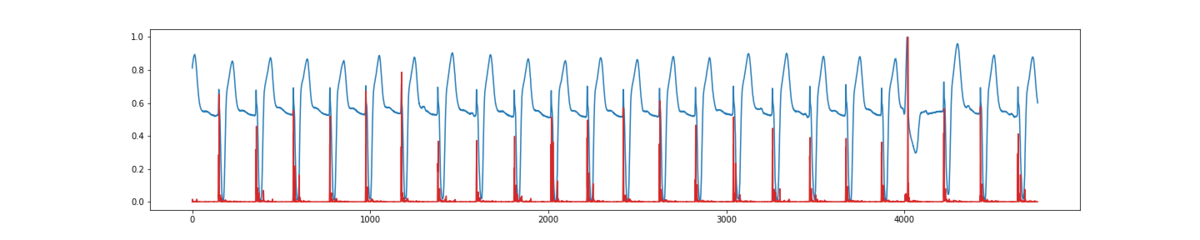
マハラノビス距離などもありますが、ここではユークリッド距離で計算します。
そこそこ誤検値している感が否めませんが、異常箇所が最も距離が遠くなっていることがわかるかと思います。
# 予測誤差の計算 dist = test_y - test_pred[:,0] u_dist = pow(dist, 2) u_dist = u_dist / np.max(u_dist) plt.figure(figsize=(20,4)) plt.plot(test_y) plt.plot(u_dist, color='tab:red') plt.show()
考察
心電図データに関してにはなりますが、オートエンコーダよりもLSTMの方が、明らかに精度高く予測できていることがわかりました。
しかし、異常検知で考えた場合、今回異常箇所として指定した場所のみが、うまく予測できない方が好ましい結果と言えます。
その点で言えば、オートエンコーダは異常箇所が極端に予測できなくなっているため、優れているように見えます(他のデータだと結果は変わるかもしれません)。
オートエンコーダとLSTMを組み合わせたモデルを作れば、さらに精度高く異常検知できるかもしれません。
スパイクの激しい心電図のデータはスパイクの頂点が正確に予測できていないため、LSTMは異常箇所以外も距離が遠くなって、誤検知が多くなったといえます。
これは生の心電図データだけでなく、他の特徴量を作り、それをLSTMに通せば改善されるような気もしています。
オートエンコーダによる再構成誤差とLSTMによる予測誤差を用いて異常検知する場合、どちらが良いかどうかの、メリット・デメリットは一概に言えないのが個人的見解です。
その点に関して、論文、書籍等で言及されているものがある場合、ぜひ一読したいですね。
参考文献
LSTMをよりわかりやすく&より詳細に!<br>(前処理・世界観の説明編) - AI学習者・実務家の「疑問」を解消したい
深層学習を用いた時系列データにおける異常検知 | Kabuku Developers Blog